Відмінності мікробіомів кишечника між диким та чорним носорогом у полоні - наслідки для здоров’я носорога
Предмети
Анотація
Вступ
З більш ніж 100 000 вільно вигулюваних африканських чорних носорогів у 1960-х роках цей вид, що перебуває під загрозою зникнення, зменшився більш ніж на 90% до приблизно 5000 тварин сьогодні 1. У середньому понад 1000 носорогів щороку браконьєрується в країнах ареалу, серед яких Південна Африка, Намібія, Кенія та Зімбабве 1. В даний час в зоологічних закладах Північної Америки мешкає менше 100 чорних носорогів (південний та східний підвид разом узятих) як резервуар проти потенційного зникнення 2. Однак ex situ населення відчуває власні загрози виживанню, включаючи безліч незвичних синдромів хвороб, які зазвичай не описані в дикій природі 3,4,5,6,7,8,9,10, а також погане відтворення 11,12 та фрагментованість популяцій 3. Нещодавні дослідження серед ссавців припустили, що мікробіомічні відмінності між дикими популяціями та популяціями, що перебувають у неволі, можуть впливати на загальний стан здоров'я в цілому, а також на функції травлення та імунітету, зокрема 4 .
Результати
Мікробіом кишечника
Прочитайте підходи до картографування та вилучення
Всі вибірки були послідовно розміщені на глибині щонайменше 6,9 мільйона зчитувань парного кінця на зразок, в середньому 12 479 613 зчитування парних кінців. Дуже мало прочитаних даних було відкинуто під час якісного обрізання (3,16%); середній показник якості в середньому становив 12 085 574 зчитування парних кінцівок на зразок.
Загалом, низькі показники картографування (Таблиця 1 Середній відсоток відображення для всіх програмних платформ для метагеномічного картографування.
Багатство оперативної таксономічної одиниці (OTU) не відповідало зразкам і на нього впливав використаний набір для вилучення. Зразки, видобуті ZymoBIOMICS, мали в середньому більше звернень OTU порівняно із зразками MoBio PowerFecal, незалежно від походження (дикий проти неволі; p = 0,099). PathoScope призначив більшу частку зчитувань набору диких зразків (R2-R11), які зберігалися в екрані Zymo DNA/RNA і витягувались за допомогою набору ZymoBIOMICS DNA Miniprep (в середньому 447354 зчитування), ніж дикі зразки, заморожені та екстраговані MoBio PowerFecal комплект (у середньому 155 729 читань). Дикі зразки Zymo мали в середньому 350 OTU на видовому рівні, тоді як дикі зразки MoBio мали 250 OTU (p = 0,036). Таким чином, метод збору впливав на склад мікробіомів. Спостережувані спільноти помітно різнилися між екстрагованими MoBio та Zymo екстрактами зразків (PERMANOVA, p Рисунок 1
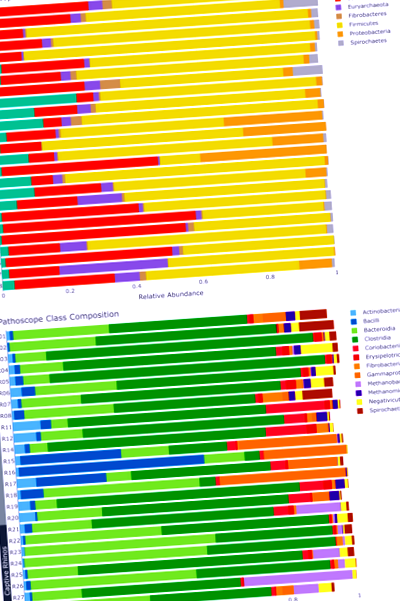
Склад мікробіома носорога, визначений PathoScope, розбитий за (A) тип і (B) класу, згрупованого за диким та захопленим хостом. Порожній простір являє собою прочитання бактерій, не ідентифікованих у відповідному таксономічному рангу. Такса, що становить в середньому менше 1% зчитувань та менше 5% у всіх зразках, була відфільтрована для візуалізації.
На рівні класу та класу кількість кількох бактерій відрізняє мікробіоми носорогів на основі дикого та неволі. Аналіз на рівні роду виявив більші відмінності між дикими та полоненими носорогами (рис. 2А). Роди Ешерихії, Осцилібактер, Псевдобутирівібріо, і Трепонема були вищими у диких носорогів, тоді як Бактероїди і Превотелла були збільшені у всіх неволі. Однак як дикі, так і носороги, що перебувають у неволі, експресували основні групи мікробів для травлення (целюлолітичні, амілолітичні), але були представлені різними видами (тобто, функціонально подібними, але таксономічно різними мікробіомами; рис. 2Б). Існував також ряд видів, котрі по-різному були поширені між дикими та носорогами в полоні. Наприклад, бактерії, що продукують метан Methanocorpusculum bavaricum (р = 0,0059) було більше у диких носорогів, тоді як у носорогах, що перебувають у полоні Methanobrevibacter ruminantium (p = 2,06e-28). Bacteroides fragilis (p = 0,0005), Steptococcus suis (p = 3,94e-15), і кишкова паличка (p Рисунок 2
Склад бактеріального мікробіома чорного носорога, визначений PathoScope, розбитий за (A) рід і (B), згруповані за дикими проти хазяїнів, що перебувають у неволі. Порожній простір містить бактеріальні показники, не ідентифіковані у відповідному таксономічному рангу. Такса, що становить в середньому менше 1% зчитувань та менше 5% у всіх зразках, була відфільтрована заради візуалізації.
Звичайні таксони були знайдені серед диких та носорогів у полоні відповідно, що свідчить про основний мікробіом носорога на основі статусу полону (Додаткова таблиця S1a, b). Тип Firmicutes домінував у мікробіомі як диких, так і полонених носорогів, що становило 32,7% та 20,8% від загальної кількості відтворених зображень та 51,1% та 48,0% основного мікробіома, відповідно. Однак наступним провідним домінуючим типом у диких носорогів були протеобактерії (23,6%) та Bacteroidetes (17,6%), тоді як Bacteroidetes були другим домінуючим типом у мікробіомі у полоні носорога (42,4%).
Хоча таксономічний склад носорогів показує відмінності між дикими мікробами та кишечником у неволі, показники альфа-різноманітності між цими двома групами були подібними. Однак спостережуване видове багатство (р = 0,082) вказувало на те, що дикі носороги мають вищу медіану спостережуваного багатства ОТУ (
335) порівняно з полоненими (
220) (рис. 3), що узгоджується з усіма таксономічними результатами. Індекси різноманітності як Шеннон (p = 0,36), так і Сімпсон (p = 0,69) вказують на те, що носороги демонструють високу різноманітність, незалежно від їх походження (дикі проти неволі), а дикі носороги демонструють дещо (але не суттєво) вищу різноманітність (рис. 3). Для всіх трьох показників альфа-різноманітності зразки, отримані від диких носорогів, демонстрували більшу дисперсію разом із більшою кількістю відхилень порівняно із зразками, що перебувають у неволі, та згруповані разом залежно від їх походження (дикий проти неволі; Рис. 4). Ці закономірності відповідали як індексам Жаккарда, так і Брея-Кертіса. Крім того, ці закономірності узгоджувались між результатами PathoScope (рис. 4A, B) та результатами PhyloSift (рис. 4C, D). Показники відмінності, виміряні окремо за індексами Брея-Кертіса, Жаккарда та Дженсена-Шеннона (JSD) з 10000 перестановок, були дуже значущими (PERMANOVA, p = 9,999e-05), репрезентативні для центроїдів, що відрізняються між двома групами . Це свідчить про те, що дві групи мають різні та різні спільноти. Ділянки ординації разом з ПЕРМАНОВОЮ показали, що мікробіоми носорогів були більш схожими на інших носорогів з однаковим статусом полону, причому дикі носороги демонструють значно більші варіації та ареал, ніж носороги в полоні.
Багатство та різноманітність OTU на рівні вибірки PathoScope (індекси Шеннона та Сімпсона) диких та носорогів у полоні.
Неметричні багатовимірні масштабні графіки даних PathoScope з використанням відстаней Жаккарда (A) та відстані Брея-Кертіса (B) та даних PhyloSift з використанням відстані Жакарда (C.) та відстані Брея-Кертіса (D).
Функціональний аналіз
Всього виявлено, що 39 термінів генної онтології (GO) диференційовано містять мікробіоми диких та неволі носорогів зі значенням Q менше 0,05 (Додаткова таблиця S2). Більшість термінів GO позитивно пов’язані з мікробіомами у полоні носорогів. Загалом 127 шляхів було диференційовано рясним, проте лише два шляхи (PWY_5103 L_isoleucine_biosynthesis_III та PWY_6121 5_aminoimidazole_ribonucleotide_biosynthesis_I) мали Q-значення нижче 0,05, ймовірно, через малий обсяг вибірки (Додаткова таблиця S3). Схоже, мікробіоми носорога, що перебувають у полоні, мають вищу активність щодо розмноження бактерій та вироблення амінокислот. Крім того, функціональні шляхи та терміни GO вказують на більш високу доступність крохмалю у полонених носорогів. Дикі та полонені носороги мають різні шляхи, що свідчить про те, що між цими двома групами відбувається різна метаболічна активність.
Обговорення
Мікробіом кишечника відіграє ключову роль у здоров’ї та самопочутті тварин, проте немає єдиної думки щодо того, як мікробіом кишечника може змінюватися між дикими тваринами та тваринами, що перебувають у неволі 4. У рослиноїдних популяція бактерій у кишечнику бере участь у розщепленні волокнистої рослинної сировини на різні метаболіти, включаючи жирні кислоти з дрібними ланцюгами (SCFA), які роблять значний вплив на здоров’я господаря. Попередні дослідження різноманітності мікробіомів кишечника у диких тварин порівняно з тваринами, що перебувають у неволі, обмежили свій висновок одним геном для ідентифікації відомих бактеріальних таксонів, якому бракує геномної ширини та таксономічної глибини, доступних завдяки метагеноміці дробовиків. Тут ми використовуємо потужний метагеномічний підхід для характеристики та тестування на відмінності в альфа-різноманітності, бета-різноманітності та функціональному різноманітті мікробіому кишечника (фекалій) диких та чорних носорогів у полоні, видів, що перебувають у критичному зникненні, з метою використання цієї інформації для оздоровлення тварин у неволі.
Висока частка бактерій, ідентифікованих у зразках калу, була збережена між дикими та носорогами, що перебувають у неволі, але також були виявлені відмінності, що відрізняли значну зміну мікробних спільнот через неволю, що також було виявлено в ряді інших досліджень у неволі, що використовували послідовність ампліконів 16S на ссавцях 5,13, 14,15,16,17. Однак наш метагеномічний підхід призвів до великої кількості непридатних для читання послідовностей (
90%) від чорних носорогів, що свідчить про відсутність відповідних та відомих геномів бактерій у базі даних. Однією з найбільших переваг метагеномічного підходу є те, що ви можете виявити та кількісно визначити невідомі мікроби, а також відомі, і наші результати вказують на те, що у цільовому секвенуванні ампліконів 16S бракує значної частини мікробного різноманіття, враховуючи те, що 90% зчитувань не можуть бути зіставлені з еталонними геномами. Цей результат був перевірений на трьох різних програмних платформах для характеристики різноманітності мікробіомів, а саме PathoScope, Kraken та Centrifuge. Через значну відсутність курованих та перевірених геном мікробів з дикої природи в геномних базах даних (тобто RefSeq NCBI), існує критична потреба у дослідженні цих недостатньо вивчених систем (тобто дикої природи та сезонної динаміки їх різноманітних мікробіомів) для реконструкції нові геноми для повної ідентифікації організмів, що присутні в їх мікробіомі. Виявлення невідомих мікробів з мікробіомів дикої природи забезпечить всі дослідницькі та медичні спільноти дикої природи необхідною інформацією для точної характеристики та потенційних змін мікробіомів видів, що перебувають у неволі, для покращення здоров’я.
Хоча ми не виявили суттєвої різниці в альфа-різноманітності мікробних спільнот між дикими популяціями чорного носорога та неволі (обидва мали велику кількість мікробних видів), бета-різноманітність було суттєво різним, припускаючи, що існують різні мікробні спільноти у диких проти чорних носорогів у полоні. . Наші результати показали збільшення розподілу мікробних показників у зразках, що перебувають у полоні, до таксонів бактерій, традиційно асоційованих з кишками жуйних тварин (таких як Ruminococcus albus, Selenomonas bovis, і Treponema bryantii), припускаючи, що невідомі (прокаріотичні геноми, яких немає у RefSeq NCBI), кишкові мікроби диких носорогів замінюються тими, що зустрічаються у звичайних домашніх тварин, одомашнених людиною. Ця заміна могла бути частково пов’язана з носорогами, які отримують дієту, подібну до корів та коней, або людьми, з якими носороги, що перебувають у полоні, часто контактують. Дикі носороги, схоже, дотримуються профілю мікробіомів ближче до здорових домашніх тварин, з більшим бета-різноманіттям, функціональним різноманіттям та варіацією між окремими носорогами порівняно з носорогами-полонями .
За дуже небагатьох досліджень мікробіома носорогів, їх найближчого домашнього родича, коней, можна використовувати як джерело порівняння. Попередні дослідження на здорових конях показали, що Firmicutes спостерігається у вищому співвідношенні порівняно з Bacteroidetes 22, тоді як більша частка Bacteriodetes пов'язана з колітом 23. Однак існує мінімальна інформація про частоту колітів у носорогів, що перебувають у полоні, і вимагає подальшого розслідування. На відміну від них, протеобактерії становили друге місце за поширеністю у дикій природі. Незважаючи на те, що протеобактерії вважаються основним мікробом рослиноїдних тварин 24, цей тип також включає широкий спектр добре відомих патогенів, таких як Кишкова паличка, Сальмонели, Вібріон, Гелікобактор та інші 25. На ці висновки може вплинути той факт, що в дикій природі носороги мають спільні джерела води (водяні ями), які часто відвідують численні інші види. Нерідкі випадки, коли тварини випорожнюються в цих районах і, як наслідок, забруднюють воду різними іншими мікробами, які в свою чергу можуть закріпитися в кишечнику тварин, що споживають цю воду.
Порівняння функціональної різноманітності мікробіома чорного носорога продемонструвало більшу кількість шляхів гліколізу та синтезу амінокислот у неволі порівняно з їх дикими аналогами, що свідчить про дисбіоз, що виникає в результаті дієти, що пропонується в неволі. Чорні носороги в полоні також свідчили про високу доступність крохмалю. Дієта носорогів у полоні складається з
36% нейтрального миючого волокна (NDF) і
25% кислотного миючого волокна (ADF) у комерційних продуктах, 36–50% NDF та 28–39% ADF у сіні люцерни та 49–69% NDF та 31–41% ADF у сіні трави 26. Найбільша частка раціону припадає на сіно з люцерни та комерційні продукти, що становить нижчий діапазон вмісту клітковини, ніж те, що спостерігали дикі носороги, що містять NDF в межах 30–78% та ADF в межах 14–59% 27. Таким чином, у порівнянні з побратимами, мікробіом диких чорних носорогів містив більшу частку бактерій, що беруть участь у розщепленні рослинної сировини. Зокрема, ми виявили більшу частку Ешерихії, Осцилібактер, Псевдобутирівібріо, і Трепонема у диких чорних носорогів. Відомо, що всі ці таксони беруть участь у розщепленні волокон. Крім того, Псевдобутирівібріо беруть участь у виробництві бутирату, який, як повідомляється, також є вищим у здорових тварин завдяки підтримці здорових сосочків у кишечнику 28. Ацетат, бутират та пропіонат SCFA мають важливе значення в ряді фізіологічних аспектів засвоєння поживних речовин, імунної функції, сигналізації клітин та захисту патогенів 29. .
Матеріали і методи
Заява про використання тварин
Усі тварини були умовно відібрані під час звичайних транслокаційних зусиль у Південній Африці або під час планових оцінок стану здоров'я в США. Отже, не потрібно було затвердження інституційного догляду та використання тварин.
Колекція
Вісім полонених зразків калу зберігали замороженими (-80 ° C) до вилучення ДНК. Вісім із 17 диких зразків були перевезені до Університету Стелленбоша в Південній Африці для вилучення ДНК і відправлені до США у вигляді очищеної геномної ДНК. Для решти дев’яти зразків диких фекалій між 1-2 грамами калу зберігали в ДНК/РНК-щит (Zymo Research, США) і транспортували до США. Ці зразки також зберігали замороженими (-80 ° C) до спроби вилучення ДНК.
Вилучення ДНК та метагеномне секвенування
Біоінформаційний аналіз
Доступність даних
Дані послідовності наступного покоління, асоційовані з цим дослідженням, були депоновані в GenBank під вступом до SRA: PRJNA532626.
- Їжте дико - зустрічайте Джо
- Дієтичні впливи на метанефрини плазми та сечі, що мають значення для діагностики
- Харчові звички, жир в організмі пов’язані з різницею в хімії мозку Національний інститут здоров’я
- Сирна чорна квасоля та рисові рецепти їжі
- Розкрито прикордонний мульти-омічний аналіз мікробіому та метаболому у здорових суб'єктів