Весь секвенування геному етнічного Патана (Пахтун) з північного заходу Пакистану
Анотація
Передумови
Пакистан охоплює ключову географічну область в історії людства, будучи одночасно частиною району річки Інд, який виступав як одна з колисок цивілізації та як сполучна ланка між Західною Євразією та Східною Азією. Цей регіон населений низкою різних етнічних груп, найбільшими з яких є панджабі, патан (пахтуни), сіндхі та белудж.
Результати
Ми проаналізували перший етнічний чоловічий геном Патана шляхом секвенування його до 29,7-кратного охоплення за допомогою платформи Illumina HiSeq2000. Загалом було виявлено 3,8 мільйона однонуклеотидних варіацій (SNV) та 0,5 мільйона малих інделів шляхом порівняння з референтним геномом людини. Серед SNV 129 441 були новими, а 10 535 несинонімічних SNV було виявлено в 5344 генах. SNV були позначені щодо наслідків для здоров'я та захворювань високого ризику, а також можливого впливу на ефективність наркотиків. Ми підтвердили, що представлений тут геном Патана є представником цієї етнічної групи, порівнявши його з панеллю середньоазіатських країн із панелей HGDP-CEPH, набраних для
650 к ОНП. МтДНК (H2) та гаплогрупа Y (L1) цієї людини також були типовими для його географічного регіону походження. Нарешті, ми реконструюємо демографічну історію PSMC, яка підкреслює нещодавнє збільшення ефективного чисельності популяції, сумісне з домішкою між європейськими та азіатськими лініями, що очікується в цьому географічному регіоні.
Висновки
Ми представляємо послідовність цілого геному та аналізи етнічного патана з північно-західної провінції Пакистану. Це корисний ресурс для розуміння генетичних змін та міграції людей по всьому азіатському континенту.
Передумови
Технологія секвенування швидко вдосконалюється, різко зменшуючи її витрати [1]. Ці швидкі досягнення значно розширили наше розуміння генетичного різноманіття людини та історії популяції [2], дозволивши нам дослідити варіанти із наслідками для здоров’я та проклавши шлях до персоналізованої медицини [3]. Дослідження асоціацій із широким асоціацією геномів (GWAS) характеризують функцію тисяч загальних SNV, але все ще залишаються незвіданими мільйони варіантів [4]. Отже, для детального вивчення рідкісних геномних варіантів необхідне секвенування цілого генома. Ряд міжнародних консорціумів розпочали послідовність послідовностей цілих геномів великих панелей, включаючи Проект 1000 геномів (www.1000genomes.org), Проект особистого геному (www.personalgenomes.org) та 100 малайських геномів [5]. Ці консорціуми, а також кілька географічно більш обмежених проектів мають на меті зрозуміти функціональні аспекти як загальних, так і унікальних варіантів у людей. У майбутньому можна очікувати, що всі окремі етнічні групи будуть секвенувати свої геноми.
Пакистан лежить на стику індійського субконтиненту на Сході, держав Центральної Азії на Заході та Китаю на його півночі. Він має унікальну соціально-релігійно-культурну історію, крім ряду етнічних та мовних груп, таких як панджабі, Патан (Пахтуни), Сіндхі та Белух (Додатковий файл 1: Рисунок S1) [6]. Хоча низка цих груп була включена до генетичних панелей, що друкують мікросателіти та SNP [7], до цього часу було проведено секвенування лише однієї особини пакистанського чоловіка невідомого етнічного походження (додатковий файл 1: Рисунок S2) [8]. Тут ми повідомляємо про першу послідовність цілого геному та аналіз самця Патана (громадянин Пакистану). Геномні варіації, включаючи однонуклеотидні варіації (SNV), невеликі вставки та делеції (indels) та області варіації кількості копій (CNVR) були виявлені шляхом вирівнювання послідовності генома Pathan до людського референтного геному (hg19). Потім варіанти коментували і сканували на відповідні функції, а також SNV, які могли модулювати реакцію на препарат. Можливі шкідливі несинонімічні SNV (nsSNV) досліджували на предмет потенційного впливу на фармакокінетику та фармакодинаміку лікарських засобів. Крім того, для оцінки впливу внесків предків у геном Патана (PTN) використовувались численні аналітичні підходи.
Результати і обговорення
Послідовність геномів та ідентифікація варіантів
ДНК, витягнуту з крові, секвенували з парним зчитуванням 90 п.н., використовуючи секвенсор Illumina HiSeq2000, даючи 1 069 127 677 зчитувань. Загалом було створено 83,3 Гб послідовностей та вирівняно до еталонного геному людини (без Ns, 2 861 343 702 п.н.), що охоплює 98,2% еталонного геному на середній глибині 28,5 × (Додатковий файл 2: Таблиця S1).
Ми виявили загалом 3 813 440 SNV, з яких 3 683 999 (96,6%) було зареєстровано в базі даних dbSNP [9], а 129 441 було новим (Таблиця 1), які далі порівнювали з кількістю нових варіантів інших окремих геномів з літератури 1: Рисунок S3) [10-19]. Існувало 1 272 912 гомозиготних та 2 540 528 гетерозиготних SNV. Всього було виявлено 18 547 SNV в кодуючих областях послідовності ДНК (CDS), 25 481 в 3 'неперекладених областях (UTR) та 4969 в 5' UTR. Всього 10315 SNV у 5344 генах були несинонімами (nsSNV).
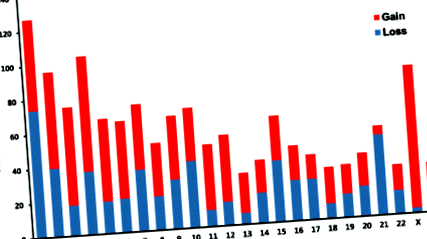
Всього було спостерігано 504 276 коротких інделів (до ± 20 основ), з них 306 128 виявлено в міжгенних регіонах, 237 в регіонах CDS та 193 308 в інтронах. Крім того, було виявлено 1503 CNVR, 713 з яких класифіковано як дубльовані, а 790 як видалені, що впливає на 2364 гени, що перекриваються (додатковий файл 3: Таблиця S2). Загалом 65 CNVR раніше не описувались у базі даних геномних варіантів (DGV; http://projects.tcag.ca/variation/). На малюнку 1 показано кількість отриманих і втрачених CNVR в кожній хромосомі. ANNOVAR був використаний для детального анотаційного аналізу CNVR для ідентифікації генів, асоційованих з цими регіонами (додатковий файл 4: Таблиця S3).
Скопіюйте області варіації чисел у геномі Патана. Кількість варіацій кількості копій, розподілена в кожній хромосомі.
Функціональна класифікація та клінічна значимість варіантів
Всі 10315 nsSNV, знайдені в геномі Патана, були додатково перевірені на предмет можливих функціональних ефектів за допомогою обчислювальних методів прогнозування (SIFT та Polyphen2), в результаті чого 43 nsSNV у 43 генах були класифіковані як функціонально пошкоджуючі (Додатковий файл 5: Таблиця S4). Крім того, nsSNV були анотовані за допомогою ClinVar для їх клінічної значущості, і ми виявили, що 31 кодує SNVs асоційовано з декількома захворюваннями (Додатковий файл 6: Таблиця S5). Особливої уваги заслуговує SNV (rs1049296, Pro570Ser) у TF ген [20], який впливає на сприйнятливість до Альцгеймера; Ser217Leu в ELAC2 ген (rs4792311), який причетний до генетичної сприйнятливості до спадкового раку простати [21]. Рівень простати низький у Пакистані (3,8%) [22], порівняно з американцями та кавказцями [23]. Три кодування SNV GHRLOS (rs696217, Leu72Met), СЕРПІН1 (rs6092, Ala15Thr) та PPARG (rs1801282, Pro12Ala), які всі пов’язані з ожирінням [24-26]. Повідомляється, що близько 22,2% пакистанців страждають ожирінням, що є близьким до європейського (
24%) та населення США (
Ми також виявили три патогенні SNV в генах, пов'язаних з волоссям, шкірою та пігментацією: ЕДАР (rs3827760, Val370Ala), SLC45A2 (rs16891982, Phe374Leu) та TYR (rs1042602, Ser192Tyr) [30-32]. Крім того, ми виявили SNV (rs17822931, Gly180Arg) у ABCC11, який відповідає за вологу вушну сірку, яка також була знайдена в пакистанському геномі PK1 [33].
Один із варіантів (rs1065852, Pro34Ser) у CYP2D6 ген відповідає за поганий метаболізм дебризохіну, адреноблокуючого препарату, що використовується для лікування гіпертонії [34]. Крім того, два SNV в TPMT (rs1142345, Tyr240Cys та rs1800460, Ala154Thr), як відомо, мають патогенну дію та призводять до дефіциту тіопурину метилтрансферази (TPMT) [35,36]. Крім того, два nsSNV (RS2056899 та RS140980900) CYP4A22 і GGT5 були знайдені гени на шляху метаболізму арахідонової кислоти (Додатковий файл 7: Таблиця S6). Арахідонова кислота в організмі людини зазвичай надходить з дієтичних тваринних джерел, таких як м’ясо, яйця та молочні продукти. М’ясо є важливою складовою дієти Патана, яке зазвичай вживають принаймні раз на день, часто у формі кабабу (смаженого в олії фаршу) або каррі [37].
Порівняльний геномний аналіз був проведений з використанням геному Патана (PTN) та іншого раніше опублікованого пакистанського генома (PK1). Несинонімічні варіанти геному Пакистану (PK1) були анотовані для дослідження супутніх захворювань. З
8000 nsSNV виявлено лише 37 варіантів (три нові), пов’язаних з певними розладами. Було виявлено вісім клінічно значущих СНВ, які перекривались геномом Патана (PTN). Ми не виявили жодних пошкоджених варіантів, що відповідають за хворобу Альцгеймера, ожиріння та хвороби серця, як у геномі Патана (PTN). SNV (rs1057910; CYP2C9) спостерігався в геномі PK1, який відомий реакцією на вафарин. Більше того, патогенна мутація (rs1169305) була помічена у HNF1A ген, який може стати причиною діабету у особи PK1.
Більшість клінічно значущих варіантів, прийнятих у цьому дослідженні, спочатку були описані в популяціях Кавказу. Хоча цей результат може бути наслідком геномних спорідненостей генома Патана з іншими кавказькими популяціями, він також може відображати упередженість через більшість робіт GWAS, що проводяться над кавказькими популяціями [38]. Тому для аутентифікації буде потрібно когортне дослідження серед пакистанського населення.
Фармакогеномічний аналіз
Порівняння з іншими особами Патана
Ми дослідили, наскільки репрезентативним був наш геном Патана для цієї етнічної групи, порівнявши його з іншими двадцятьма двома особами Патана в панелі HGDP-CEPH [7], яку було введено для
650 к SNV, разом з ще 190 особами з інших восьми популяцій Південної Азії (Пакистану) з тієї ж групи. Аналіз суміші проводили на основі 643 281 SNV (розріджених, щоб уникнути LD). Ми розглянули приналежність кластера до STRUCTURE (від K = 2 до K = 5), склад генома Pathan (PTN) знаходився в межах мінливості, яка спостерігалась у вибірці Pathan з HGDP (рис. 2). Подібним чином, на графіку багатовимірного масштабування (MDS) геном Патана потрапляв до інших особин Патана (Додатковий файл 1: Рисунок S4). У сукупності ці два результати підтверджують, що геном Патана, представлений у цій роботі, є представником етнічної групи Патан. Ці результати також узгоджуються із власним походженням суб'єкта, коли всі його бабусі та дідусі приїжджали з Афганістану в Хайбер-Пахтунхва (Пакистан).
Результати змішування особи Патана (PTN) з іншими етнічними групами в Південній Азії. Результати суміші для K = 2 та K = 5 для особини Патана в поєднанні з вісьма етнічними геномами з набору даних HGDP. Аналіз базувався на 643 281 SNV. Кожна особа представлена вертикальною лінією, розділеною на кольорові сегменти, що представляють коефіцієнти належності в підгрупах.
аналізи мтДНК та Y-хромосом
Повний мітохондріальний геном особини Патана був сформований шляхом зіставлення його зчитувань із оновленою послідовністю Кембриджа (rCRS) [45]. Вміст аденину та тиміну (AT) у геномі становив 55,5%, тоді як вміст гуаніну та цитозину (GC) становив 44,5%. Всього в мітохондріальному геномі Патана виявлено 57 SNV, про 13 з яких раніше не повідомлялося. Потім варіанти були зіставлені з MitoVariome [46] для ідентифікації мітохондріальної гаплогрупи нашого індивіда Pathan. Загалом 14 SNV діагностували гаплогрупу Н2, яка, як стверджується, має ексклюзивне кавказьке походження, і її граничне поширення у Патанах відображає домішки [47].
Вміст AT та GC Y-хромосоми становив 39,87% та 60,13% відповідно. Всього було виявлено 13 724 SNV, з них 4423 нові. Спостережувані Y-хромосомні SNV були позначені як маркери для гаплотипу L1 клади L. Гаплогрупа L має високу частоту в Пакистані (14%) порівняно з Індією (6,3%), Туреччиною (
4%) та кавказці (
Аналіз демографічної історії
Ми зробили висновок про демографічну історію Патана, використовуючи послідовно послідовну марківську модель коалесценції (PSMC) [51] (рис. 3), і порівняли її з групою популяцій у всьому світі на основі ряду геномів HGDP [52]. Як повідомлялося раніше, усі популяції мають схожу демографічну історію від 1 мільйона до 200 тисяч років тому. Починаючи з 200 тисяч років тому і до 20 років тому Патани рухаються за аналогічною траєкторією до інших азіатських та європейських популяцій, з висновком ефективного розміру популяції менше, ніж африканських, що відображає вузьке місце поза Африкою. Протягом останніх 20 тис. Років Патан демонструє вибух ефективного розміру популяції, одночасний з іншими євразійськими популяціями, але значно більший за масштабами. Дуже великий ефективний розмір популяції, швидше за все, відображає домішку між європейськими та азіатськими лініями, що породжує сучасних патанців (як це також припускає аналіз мтДНК та Y-хромосоми), а не фактичне збільшення чисельності перепису.
Висновки про історичну чисельність популяції шляхом парного послідовного аналізу марковійського коалесценту. Аналіз PSMC (Pairwise Sequentially Markovian Coalescent) був проведений для реконструкції демографічної історії популяції Патан, порівняно з набором з 11 геномів HGDP з усього світу (Африка: Дінка, Йоруба, Манденка, Мбуті, Сан; Азія: Дай, Хань; Європа: французька, сардинська; Океанія: папуаська; Америка: Карітіана).
Висновки
Тут ми вперше представляємо цілий геном особини Патана з північно-західної провінції (Хайбер-Пахтунхва) Пакистану. Наш аналіз забезпечує детальний огляд різноманітності геному Патана та функціональної класифікації варіантів та його впливу на фармакогеноміку. Потрібен широкомасштабний аналіз різноманітних геномів, щоб допомогти дослідникам у всьому світі зрозуміти генетичне різноманіття та функціональну класифікацію варіантів, а також фармакогеномічні ознаки та пов'язані з ними препарати, які можна використовувати як персоналізовану медицину.
Методи
Вибір теми та етичне висловлювання
Це дослідження було проведено відповідно до Гельсінської декларації та схвалено Фондом досліджень геному (GRF) Організаційної комісії з IRB-REC-2011-10-003. Підписані поінформовані згоди були отримані від учасника цього дослідження та згоди членів його родини на публікацію всього вмісту інформації про геном та фенотип, а також особисту ідентифікаційну інформацію (таку як вік, стать та місцезнаходження).
Є задокументовані випадки, коли члени його сім'ї страждали на гіпертонію, проблеми з серцем, неврологічними розладами, діабетом та ожирінням. Його батькові поставили діагноз: серцево-судинні розлади, гіпертонія та хвороба Альцгеймера. Його мати страждає артрозом, а бабуся і дідусь померли через серцевий напад, рак та гіпертонію.
Джерела даних
Довідковий геном UCSC (hg19, лютий 2009 р.), Версія dbSNP 137 та анотації генома були завантажені з бази даних (www.genome.ucsc.edu). Геноми з панелі HGDP-CEPH, що складається з 190 особин, належать до восьми популяцій Південної Азії (Балочі, Брахуї, Бурушо, Хазара, Калаш, Макрані, Патан та Сіндхі), які були набрані для
650 к SNV були отримані з загальнодоступної бази даних.
Вилучення ДНК
Геномна ДНК була вилучена з лімфоцитів артеріальної крові тридцятирічного чоловіка Пакистану Пафана, який проживав у північно-західній провінції Пакистану. Міні-набір QIAamp DNA Blood Mini був використаний для вилучення ДНК із крові (Qiagen). Нанодроп Tecan’s Infinite F200 був використаний для оцінки чистоти ДНК, 1,7% агарозний гель-електрофорез для підтвердження розміру ДНК (наявність ДНК з високою молекулярною масою) та флуорометр Qubit Invitrogen для визначення концентрації ДНК.
Цитогенетичний аналіз
Каріотипування проводили з культивованими лімфоцитами периферичної крові із застосуванням стандартних методик, а смужку GTG використовували для ідентифікації хромосомних аберацій, що корисно для ідентифікації генетичних захворювань за допомогою фотографічного зображення всього комплементу хромосом [53]. Явних хромосомних відхилень у цитогенетичному аналізі за допомогою візуалізації хромосомної каріотипуючої G-смуги не виявлено (додатковий файл 1: Рисунок S5).
Підготовка бібліотеки та секвенування цілого генома
Дві парні кінцеві бібліотеки готували з 1,1 мкг гДНК за допомогою набору для підготовки ДНК Illumina TruSeq, дотримуючись стандартного протоколу Illumina (Набір для підготовки спарених бібліотек, Illumina, SanDiego, Каліфорнія, США). Зсув гДНК проводили з використанням серії Covaris S (Covaris, MS, USA). Після закінчення ремонту, A-хвоста та перев'язки адаптерів ДНК в діапазоні 500-600 п.н. очищали від 2% агарозного гелю. Потім ДНК збагачували ПЛР загалом десять циклів. Потім правильний розмір ДНК підтверджували за допомогою біоаналізатора Agilent, після чого проводили кількісну оцінку qPCR за допомогою реагентів Roche Light Cycler 480 II та Kapa Biosystems.
Генерація кластерів виконувалась на cBot Illumina, а бібліотеки послідовно виконувались на Illumina HiSeq 2000 за протоколом Paired-End. Доступ до послідовностей можна отримати за адресою NCBI SRA з номером приєднання SRA092047. Решту нашого аналізу було розпочато з файлів FASTQ, наданих набором програм для аналізу CASAVA компанії Illumina.
Картування та вирівнювання до еталонного геному
Послідовності геномів вирівнювали з референтним геномом людини (hg19), використовуючи Burrows-Wheeler Aligner (BWA; версія 0.5.9) [54] та SAMtools 0.1.16 [55] за замовчуванням, крім “aln -t 3 -l Варіанти 45-к 2 ”. Потім файли вирівнювання були об’єднані в один файл BAM, позначені для дублікатів за допомогою Picard 1.59 (http://picard.sourceforge.net), а базові показники якості були відкалібровані за допомогою Набору інструментів аналізу геномів (GATK v1.4) [56].
SNV, короткі індекси та CNV, що дзвонять
SNV та невеликі показники в діапазоні від 1 до 20 баз були ідентифіковані за допомогою набору інструментів аналізу генома (GATK v1.4) з HARD_TO_VALIDATE: MQ0 ≥ 4 та ((MQ0/(1,0 × DP))> 0,1), 2) QualFilter = QUAL 2 більший ніж 0,1 з будь-яким іншим SNV у тому ж вікні). Компоненти MDS були отримані за допомогою опції PLINK mds-plot на основі матриці відстані ідентичності за станом (IBS). Аналіз домішок проводили за допомогою програми СТРУКТУРА для виявлення наявності різноманітних родових зв'язків геному Патана (PTN) з іншими [63]. Ми дослідили значення K від 2 до 5 і вибрали значення K, яке дало найменшу похибку перехресної перевірки.
Попара послідовно марковіанський коалесцентний аналіз
Ми провели аналіз PSMC (Pairwise Sequentially Markovian Coalescent), щоб відновити демографічну історію населення Патанса [51]. Ми порівняли геном Патана з набором з 11 геномів HGDP з усього світу (як опублікував Meyer та ін.) [52]. Спочатку ми використовували самотули для вилучення диплоїдних геномів з їх файлів BAM, вирівняних за hg19, і виключали статеві хромосоми та мітохондріальні геноми, оскільки вони гаплоїдні. У PSMC ми використовували параметри командного рядка -N25 -t15 -r5 -p "4 + 25 * 2 + 4 + 6" які були успішно використані в попередніх аналогічних аналізах людини та мавп [64].
- Проблеми з сечею - журнал Whole Dog
- Тонко нарізаний Unicode представляє нові смайлики до їжі, Whole Foods підвищує збори постачальникам тощо;
- 9-й блог «Виклик усього життя» закінчений
- 7 здорових звичок виклику цілого життя - Фермонт Мірамар
- Що таке етнічна дегустаційна таблиця