Порівняння геномів м’ясоїдних, всеїдних та рослиноїдних ссавців з новою леопардовою збіркою
Сунок Кім
1 Відділ оцінки біологічних та генетичних ресурсів, Національний інститут біологічних ресурсів, Інчхон, 22689 Республіка Корея
Юн Сон Чо
2 Інститут геноміки, Ульсанський національний інститут науки і техніки (UNIST), Ульсан, 44919 Республіка Корея
3 Департамент біомедичної інженерії Школи наук про життя Ульсанського національного інституту науки і технологій (UNIST), Ульсан, 44919 Республіка Корея
4 Інститут особистої геноміки, Фонд досліджень геному, Чхонджу, 28160, Республіка Корея
Хак-Мін Кім
2 Інститут геноміки, Ульсанський національний інститут науки і техніки (UNIST), Ульсан, 44919 Республіка Корея
3 Департамент біомедичної інженерії Школи наук про життя Ульсанського національного інституту науки і технологій (UNIST), Ульсан, 44919 Республіка Корея
Оксунг Чунг
4 Інститут особистої геноміки, Фонд досліджень геному, Чхонджу, 28160, Республіка Корея
Хюньо Кім
5 Geromics, Ульсан, 44919 Республіка Корея
Sungwoong Jho
4 Інститут особистої геноміки, Фонд досліджень геному, Чхонджу, 28160, Республіка Корея
Гонг Соомун
6 Відділ тваринних ресурсів, Національний інститут біологічних ресурсів, Інчхон, 22689 Республіка Корея
Чонхо Кім
7 зоопарк Чеонджу, Чхонджу, 28311 Республіка Корея
Ву Юнг Банг
1 Відділ оцінки біологічних та генетичних ресурсів, Національний інститут біологічних ресурсів, Інчхон, 22689 Республіка Корея
Чанму Кім
1 Відділ оцінки біологічних та генетичних ресурсів, Національний інститут біологічних ресурсів, Інчхон, 22689 Республіка Корея
Джунгва Ан
6 Відділ тваринних ресурсів, Національний інститут біологічних ресурсів, Інчхон, 22689 Республіка Корея
Чанг Хван Бае
1 Відділ оцінки біологічних та генетичних ресурсів, Національний інститут біологічних ресурсів, Інчхон, 22689 Республіка Корея
Янгджуне Бхак
2 Інститут геноміки, Ульсанський національний інститут науки і техніки (UNIST), Ульсан, 44919 Республіка Корея
Сунвон Чон
2 Інститут геноміки, Ульсанський національний інститут науки і техніки (UNIST), Ульсан, 44919 Республіка Корея
3 Департамент біомедичної інженерії Школи наук про життя Ульсанського національного інституту науки і технологій (UNIST), Ульсан, 44919 Республіка Корея
Хьеджун Юн
2 Інститут геноміки, Ульсанський національний інститут науки і техніки (UNIST), Ульсан, 44919 Республіка Корея
3 Департамент біомедичної інженерії Школи наук про життя Ульсанського національного інституту науки і технологій (UNIST), Ульсан, 44919 Республіка Корея
Юмі Кім
2 Інститут геноміки, Ульсанський національний інститут науки і техніки (UNIST), Ульсан, 44919 Республіка Корея
JeHoon черв
4 Інститут особистої геноміки, Фонд досліджень геному, Чхонджу, 28160, Республіка Корея
5 Geromics, Ульсан, 44919 Республіка Корея
ХеДжин Лі
4 Інститут особистої геноміки, Фонд досліджень геному, Чхонджу, 28160, Республіка Корея
5 Geromics, Ульсан, 44919 Республіка Корея
Суан Чо
4 Інститут особистої геноміки, Фонд досліджень геному, Чхонджу, 28160, Республіка Корея
5 Geromics, Ульсан, 44919 Республіка Корея
Ольга Уфиркіна
8 Інститут біології та ґрунтознавства Далекосхідного відділення Російської академії наук, Владивосток, 690022 Росія
Олексій Костиря
8 Інститут біології та ґрунтознавства Далекосхідного відділення Російської академії наук, Владивосток, 690022 Росія
Джон Гудріч
9 Пантера, Нью-Йорк, Нью-Йорк 10018 США
Дейл Мікель
10 Товариство охорони дикої природи, 2300 Південний бульвар, Бронкс, Нью-Йорк 10460 США
11 Кафедра екології, Далекосхідний федеральний університет, Айякс, острів Російський, Владивосток, 690950 Росія
Мелодія Ролке
12 Програма лабораторії наук про тварин, Leídos Biomedical Research Inc., Національна лабораторія Фредеріка, Фредерік, MD 21702 США
Джон Льюїс
13 Міжнародна ветеринарна група зоопарку (Великобританія) IZVG LLP, Station House, Parkwood Street, Keighley, BD21 4NQ UK
Андрій Юрченко
14 Центр біоінформатики геному Феодосія Добжанського, Санкт-Петербурзький державний університет, Санкт-Петербург, 199004 Росія
Антон Банкевич
15 Центр алгоритмічної біотехнології, Інститут поступальної біомедицини, Санкт-Петербурзький державний університет, Санкт-Петербург, 199034 Росія
Жуок Чо
16 Широкий інститут Массачусетського технологічного університету та Гарварду, Кембридж, Массачусетс 02142 США
Семін Лі
2 Інститут геноміки, Ульсанський національний інститут науки і техніки (UNIST), Ульсан, 44919 Республіка Корея
3 Департамент біомедичної інженерії Школи наук про життя Ульсанського національного інституту науки і технологій (UNIST), Ульсан, 44919 Республіка Корея
17 Департамент біомедичної інформатики Гарвардської медичної школи, Бостон, Массачусетс 02115 США
Джеремі С. Едвардс
18 Хімія та хімічна біологія, Комплексний онкологічний центр ООН, Університет Нью-Мексико, Альбукерке, штат Нью-Йорк 87131 США
Джессіка А. Вебер
19 Біологічний факультет Університету Нью-Мексико, Альбукерке, штат Нью-Мексико 87131 США
Джо Кук
20 Зоологічне товариство Лондона, Лондон, NW1 4RY Великобританія
Сангсу Кім
21 Кафедра біоінформатики та біологічних наук, Університет Сунсіл, Сеул, 06978 Республіка Корея
Повісити Лі
22 Банк ресурсів збереження геному корейської дикої природи, Коледж ветеринарної медицини, Сеульський національний університет, Сеул, 08826 Республіка Корея
Андреа Маніка
23 Кафедра зоології Кембриджського університету, Даунінг-стріт, Кембридж, CB2 3EJ, Великобританія
Ільбеум Лі
24 Daejeon O-World, Daejeon, 35073 Республіка Корея
Стівен Дж. О’Брайен
14 Центр біоінформатики геному Феодосія Добжанського, Санкт-Петербурзький державний університет, Санкт-Петербург, 199004 Росія
25 Океанографічний центр 8000 N. Ocean Drive, Південно-Східний університет Нова, Форт-Лодердейл, FL 33004 США
Джон Бхак
2 Інститут геноміки, Ульсанський національний інститут науки і техніки (UNIST), Ульсан, 44919 Республіка Корея
3 Департамент біомедичної інженерії Школи наук про життя Ульсанського національного інституту науки і технологій (UNIST), Ульсан, 44919 Республіка Корея
4 Інститут особистої геноміки, Фонд досліджень геному, Чхонджу, 28160, Республіка Корея
5 Geromics, Ульсан, 44919 Республіка Корея
Джо-Хон Йе
1 Відділ оцінки біологічних та генетичних ресурсів, Національний інститут біологічних ресурсів, Інчхон, 22689 Республіка Корея
Пов’язані дані
Проект цілого геномного дробовика з леопардом був депонований у DDBJ/EMBL/GenBank під вступом> LQGZ00000000. У цьому документі описана версія> LQGZ01000000. Зчитування послідовності необроблених ДНК подано в базу даних NCBI Sequence Read Archive (SRA321193). Усі дані, використані в цьому дослідженні, також доступні на ftp://biodisk.org/Distribute/Leopard/.
Анотація
Передумови
У ссавців є три основні дієтичні групи: м’ясоїдні, всеїдні та рослиноїдні. В даний час існує обмежений порівняльний аналіз геноміки щодо еволюції дієтичної спеціалізації у ссавців. Завдяки недавнім досягненням технологій секвенування, нам вдалося провести поглиблений аналіз цілого геному представників цих трьох дієтичних груп.
Результати
Ми досліджували еволюцію м'ясоїдних тварин, порівнюючи 18 представницьких геномів з усіх ссавців з м'ясоїдними, всеїдними та рослиноїдними дієтичними спеціалізаціями, зосереджуючи увагу на фелідах (домашніх котах, тиграх, левах, гепардах і леопардах), геномах гомінід та бовідів. Ми створили новий високоякісний збірний геном леопарда, а також два цілих генома дикого амурського леопарда. На додаток до чіткого скорочення в сімействах генів метаболізму крохмалю та сахарози, геноми м’ясоїдів показали свідчення спільних еволюційних адаптацій у генах, пов’язаних з дієтою, м’язовою силою, спритністю та іншими рисами, відповідальними за успішне полювання та споживання м’яса. Крім того, аналіз висококонсервативних регіонів на сімейному рівні виявив молекулярні ознаки адаптації до їжі в кожному з видів Felidae, Hominidae та Bovidae. Однак, на відміну від м’ясоїдних, всеїдні та рослиноїдні демонстрували менше спільних адаптивних ознак, що вказує на те, що м’ясоїдні піддаються сильному селективному тиску, пов’язаному з дієтою. Нарешті, феліди продемонстрували нещодавнє скорочення генетичного різноманіття, пов’язане зі зменшенням чисельності популяції, що може бути пов’язано з негнучким характером їх суворої дієти, що підкреслює їхню вразливість та критичний статус збереження.
Висновки
Наше дослідження пропонує широкомасштабний порівняльний геномний аналіз на сімейному рівні для вирішення геномних змін, пов'язаних зі спеціалізацією дієти. Наші геномні аналізи також надають корисні ресурси для генетичних досліджень, пов’язаних з дієтою.
Електронний додатковий матеріал
Інтернет-версія цієї статті (doi: 10.1186/s13059-016-1071-4) містить додаткові матеріали, доступні для авторизованих користувачів.
Передумови
Дієта - це, мабуть, найсерйозніша селекційна сила серед усіх видів на Землі. Зокрема, м’ясоїдна тварина цікава тим, що вона неодноразово еволюціонувала в ряді кладів ссавців [1, 2]. У скам’янілих копалинах спеціалізація м’ясоїдних тварин часто пов’язана з відносно короткими часами вимирання, ймовірно наслідком малого розміру популяції, пов’язаного з харчуванням у верхній частині трофічної піраміди [1, 2]. Дійсно, багато фахівців з хижаків мають близькоспоріднені види, які мають набагато ширший раціон харчування, такі як білі ведмеді, гризлі (всеїдні) та панди (рослиноїдні) в Ursidae [3, 4] та лисиці (всеїдні) у Canidae [5], підкреслюючи часту еволюційну нестабільність цього способу життя.
Тут ми досліджували геномні адаптації до дієт, спочатку розширивши геномний покрив Felidae, створивши найякісніший збірний еталонний геном великої кішки для леопарда (Panthera pardus) та цілі дані про геном для леопардової кішки (Prionailurus bengalensis). Леопарди - найпоширеніші види великих котів (від Африки до Російського Далекого Сходу), які процвітають у різноманітних середовищах [20]. Цей леопардовий збір забезпечує додатковий неодомашнений геном великої кішки, який можна спільно аналізувати з найточнішим посиланням на геном домашньої кішки, що призводить до надійних досліджень генетичної варіації геномних масштабів серед Felidae. Ці нові дані дозволили нам порівняти п'ять посилань на котів (домашня кішка, тигр, гепард, лев і леопард) і два повторно секвенуваних геноми (сніговий барс і леопардовий кіт) на рівні охоплення, порівнянному з іншими добре вивченими групами, такими як гомініди. та членистоногі. Скориставшись цим безліччю даних, ми провели ряд порівняльних аналізів для дослідження молекулярних пристосувань до м'ясоїдної.
Результати і обговорення
Секвенування та складання геному леопарда
Ми побудували еталонний геном леопарда на зразку м’яза, отриманому від самки амурського леопарда з Daejeon O-World of Korea (додатковий файл 1: Додаткові методи для деталей ідентифікації видів за допомогою аналізу генів мітохондріальної ДНК (mtDNA); додатковий файл 2: Рисунок S1). Вилучену ДНК секвенували до середньої глибини охоплення 310 × за допомогою платформ Illumina HiSeq (додатковий файл 3: Таблиці S1 та S2). Послідовні зчитування фільтрували, а потім виправляли помилки за допомогою аналізу K-mer. Розміри геному леопарда оцінювались у
2,45 Гб (Додатковий файл 1: Додаткові методи для отримання деталей; Додатковий файл 2: Рисунок S2; Додатковий файл 3: Таблиця S3). Виправлені помилки збірки були зібрані за допомогою програмного забезпечення SOAPdenovo2 [21] на 265 373 контиги (N50 довжиною 21,0 кб) і 50 400 лісів (N50 довжина 21,7 Мб), загальною довжиною 2,58 Гб (Додатковий файл 1: Додаткові методи для деталей; Додаткові файл 3: таблиця S4). Крім того, 393 866 синтетичних тривалих зчитувань Illumina TruSeq [22] (TSLR, 2,0 Гб загальної кількості баз;
Еволюційний аналіз хижих тварин порівняно з всеїдними та рослиноїдними
Щоб дослідити геномні адаптації до різних дієт та пов'язаного з ними способу життя, ми провели широке ортологічне порівняння генів серед восьми хижих (леопард, кішка, тигр, гепард, лев, білий ведмідь, косатка та тасманійський диявол), п’ять всеїдних (людина, миша, собака, свиня та опосум) та п’ять травоїдних геномів ссавців (гігантська панда, корова, кінь, кролик та слон; Додатковий файл 1: Додаткові методи для отримання детальної інформації про критерії вибору видів; Додатковий файл 3: Таблиця S13). Ці порівняння виявили численні генетичні ознаки, що відповідають молекулярним адаптаціям до гіпержадливого способу життя.
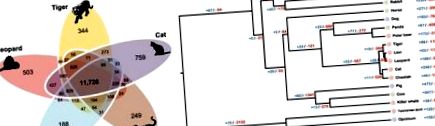
Зв'язок Felidae з іншими видами ссавців. a Ортологічні скупчення генів у видів Felidae. Ортологічні кластери генів були побудовані з використанням 18 геномів ссавців. На цьому малюнку показано лише кластери генів видів Felidae. b Розширення або скорочення генів у видів ссавців. Номери гілок вказують на кількість сімейств генів, які розширилися (сині) та скоротились (червоні) після відриву від загального предка. Кольори кіл представляють дієтичні групи (світло-червоний: хижак, світло-блакитний: всеїдна тварина, світло-зелений: рослиноїдна тварина). Лінії часу вказують час розбіжностей між видами
Еволюція копій генів та зміни амінокислот (AAC) у листяних та хижих тварин. a Контрактні (UGT1 та UGT2) та розширені (UGT3) сімейства UDP-глюкуроносилтрансферази у хижих тварин. Червоні, фіолетові, сині та чорні вузли - це гени сімейства UGT у п’яти котів, не котячих м’ясоїдних (білий ведмідь, косатка та тасманійський диявол), п’яти травоїдних та п’яти всеїдних, відповідно. b Збіжна AAC виявлена у хижих тварин. У верхній частині зображено ген людського зародка (EMB) та передбачувані білкові структури. Амінокислоти, специфічні для м'ясоїдних тварин (269-й залишок у білку ЕМВ людини, трансмембранна область) та тканини (309-й залишок, цитоплазматична область) у білку ЕМВ, показані червоним та жовтим відповідно. Цифри в дужках - це кількість геномів, проаналізованих у цьому дослідженні
Відомо, що коти не мають здатності синтезувати достатню кількість вітаміну А та арахідонової кислоти, що робить їх необхідними [30]. Цікаво, що гени сімейства цитохрому P450 (CYP), які беруть участь у катаболізмі ретинолу/лінолевої кислоти/арахідонової кислоти, зазвичай отримували контракти у всіх м’ясоїдних дієтичних групах (Felidae, порядок м’ясоїдів, косатка та тасманійський диявол; додатковий файл 3: Таблиці S18 – S29). Ретиноева кислота, перетворена з ретинолу, необхідна для ремінералізації зубів та росту кісток [31, 32], а арахідонова кислота сприяє відновленню та росту скелетної м’язової тканини після фізичних вправ [33]. Ми припускаємо, що скорочення генів сімейства CYP може допомогти м’ясоїдним тваринам утримувати на своєму тілі достатній рівень концентрації ретинолу та арахідонової кислоти, і, отже, вони могли еволюціонувати, щоб мати міцні м’язи, кістки та зуби для успішного полювання.
Ми також виявили збіжні ААК у хижих (феліди, білий ведмідь, косатка та тасманійський диявол) та травоїдних (гігантська панда, корова, кінь, кролик та слон). Лише один ген зародження (EMB) мав збіжну ААС у хижих тварин (крім тасманійського диявола), а у травоїдних не було збіжної ААС (рис. 2б), що відповідає припущенню про те, що адаптивна молекулярна збіжність, пов’язана з фенотиповою збіжністю, є рідкістю [49 ]. Цікаво, що, як відомо, ЕМВ, який, як передбачалося, буде функціонально змінений у трьох кладах хижих тварин, відіграє роль у зростанні рухових нейронів та у формуванні нервово-м’язових з’єднань [50]. Ми підтвердили, що AAC гена EMB також зберігається у домашнього тхора. Крім того, передбачалося, що 18 та 56 генів будуть специфічними для хижаків та травоїдних функцій, відповідно, зміненими принаймні одним AAC (Додатковий файл 4: Таблиці даних S7 та S8). Серед генів, змінених у специфічній для м'ясоїдів функції, відомо, що кілька генів пов'язані із скороченням м'язів (TMOD4 та SYNC) та синтезом стероїдних гормонів (STAR).
Загальнородинні регіони з високим рівнем збереження
Збереження послідовностей ДНК у видів відображає функціональні обмеження, а отже, характеристика закономірностей генетичних варіацій є критично важливою для розуміння динаміки геномних змін та відповідної адаптації кожного та групи видів [51, 52]. Ми просканували гомозиготні геномні регіони, які сильно зберігаються серед видів у межах родин: Felidae (кішка, тигр, лев, гепард, леопард, сніговий барс і леопардова кішка, час розбіжності:
15,9 мільйона років тому [MYA], хижаки), Hominidae (людина, шимпанзе, бонобо, горила та орангутанг,
15,8 MYA, всеїдні тварини) та Bovidae (корова, коза, вівця, водяний буйвол та яка,
26 MYA, травоїдні тварини) [53–55]. Ці висококонсервативні регіони (HCR) представляють зменшення генетичних варіацій (гомозиготні регіони, розподілені між видами, що належать до одного сімейства; Рис. 3 та Додатковий файл 3: Таблиці S39 та S42). Загалом було виявлено 1,13 Гб Felidae, 0,93 Гб Hominidae та 0,88 Гб HCV Bovidae із суттєво зменшеними генетичними варіаціями (скоригований P 3 та Додатковий файл 3: Таблиці S44 та S45), як очікувалося. Потім ми досліджували гени, характерні для сім’ї (1436 у Felidae, 2477 у Hominidae та 1561 у Bovidae) в HCR. Специфічні гени Felidae значно збагачені сенсорним сприйняттям світлового подразника (GO: 0050953, 27 генів, P = 0,0022), синаптичною передачею (GO: 0007268, 33 гени, P = 0,0044), передачею нервового імпульсу (GO: 0019226, 37 генів, P = 0,0054) та шлях наведення аксона (20 генів, P = 0,0054; Додатковий файл 3: Таблиці S46 та S47), що натякає на адаптацію до швидких рефлексів, виявлених у котів. Характерно, що гени, характерні для Felidae, також були функціонально збагачені для біосинтетичного процесу вуглеводів (GO: 0016051, 18 генів, P = 0,00061). Це може бути пов’язано з хижацьким режимом годівлі ніг (м’ясна дієта, тому низька дієтична кількість вуглеводів). З іншого боку, специфічні для Bovidae гени були збагачені сенсорним сприйняттям запаху (GO: 0007608, 82 гена, P = 2,44 × 10 –16) та когніцією (GO: 0050890, 113 генів, P = 2,54 × 10 –9; Додатковий файл 3: Функції таблиць S48 – S50), що вказують на пристосованість травоїдних тварин до захисних механізмів від отруєння токсичними рослинами [56].
- DK - дієта всеїдних - 15 кг Kiezebrink UK Ltd
- Щільність, напрямки або обидва Порівняння мір прохідності у зв'язку з транспортом
- Вплив дієти з низьким вмістом жиру та високим вмістом вуглеводів на збір, виготовлення та тригліцериди ЛПНЩ
- Порівняння мікробної фітази та фосфату дикальцію для росту та мінералізації кісток
- Порівняння вбудованих підключених діодних решіток та електрохімічних детекторів у високоефективних