Дискримінація між Streptococcus pneumoniae і Стрептококовий міт на основі сортування їх мас-спектрів MALDI
Науково-дослідний інститут фізико-хімічної медицини, Москва, Росія
Відповідний автор: Л. Н. Ікряннікова, Науково-дослідний інститут фізико-хімічної медицини Міністерства охорони здоров'я Російської Федерації, вул. Малая Пироговська, 119992, Москва, Росія
Науково-дослідний інститут фізико-хімічної медицини, Москва, Росія
Науково-дослідний інститут фізико-хімічної медицини, Москва, Росія
Національне агентство з клінічної фармакології та фармації, Москва, Росія
Національне агентство з клінічної фармакології та фармації, Москва, Росія
Науково-дослідний інститут фізико-хімічної медицини, Москва, Росія
Національне агентство з клінічної фармакології та фармації, Москва, Росія
Національне агентство з клінічної фармакології та фармації, Москва, Росія
Науково-дослідний інститут дитячих інфекцій, Санкт-Петербург, Росія
Санкт-Петербурзький державний медичний університет, Санкт-Петербург, Росія
Науково-дослідний інститут фізико-хімічної медицини, Москва, Росія
Науково-дослідний інститут фізико-хімічної медицини, Москва, Росія
Відповідний автор: Л. Н. Ікряннікова, Науково-дослідний інститут фізико-хімічної медицини Міністерства охорони здоров'я Російської Федерації, вул. Малая Пироговська, 119992, Москва, Росія
Науково-дослідний інститут фізико-хімічної медицини, Москва, Росія
Науково-дослідний інститут фізико-хімічної медицини, Москва, Росія
Національне агентство з клінічної фармакології та фармації, Москва, Росія
Національне агентство з клінічної фармакології та фармації, Москва, Росія
Науково-дослідний інститут фізико-хімічної медицини, Москва, Росія
Національне агентство з клінічної фармакології та фармації, Москва, Росія
Національне агентство з клінічної фармакології та фармації, Москва, Росія
Науково-дослідний інститут дитячих інфекцій, Санкт-Петербург, Росія
Санкт-Петербурзький державний медичний університет, Санкт-Петербург, Росія
Науково-дослідний інститут фізико-хімічної медицини, Москва, Росія
Анотація
Вступ
Streptococcus pneumoniae, Streptococcus mitis, Streptococcus pseudopneumoniae і Streptococcus oralis є близькоспорідненими видами трептококів групових грибів (VGS), що колонізують ротову порожнину людини; однак їх патогенні властивості суттєво відрізняються. Поки S. pneumoniae є основним людським патогеном, асоційованим із позалікарняною пневмонією, менінгітом та середнім отитом, інші представники цієї групи є коменсалами і можуть викликати інфекції лише тоді, коли вони отримують доступ до кровотоку або в умовах імунітету. Клінічні лабораторії повинні мати можливість точно диференціювати S. pneumoniae з інших VGS, які зазвичай зустрічаються в клінічних зразках для полегшення відповідної антимікробної терапії. Однак звичайні фенотипічні методи, такі як морфологія колоній, розчинність у жовчі та тестування на чутливість до оптохінів, а також комерційні системи (API 20 Strep та Vitek 2; bioMe'rieux, Marcy l'Etoile, Франція), не завжди забезпечують точну ідентифікацію. 4 .
Різні гени використовувались як мішені для дискримінації VGS на основі ПЛР: пневмолізин (курсувати) 5, автолізин (lytA) 6, поверхневий пневмококовий антиген A (psaА) 7, і фрагмент ДНК невідомої функції Spn9802 8. Однак застосування цієї стратегії ускладнюється звітами, які Стрептококовий міт і Streptococcus oralis містять гени, що кодують аутолізин та пневмолізин 9-11. Аналіз послідовності генів 16S рРНК також не можна застосовувати для дискримінації VGS через 99% схожості в нуклеотидному складі генів 16S рРНК у цих бактерій 12, 13. Аналіз послідовності rnpB (РНК-субодиниця ендонуклеази Р), содаA (марганцезалежна супероксиддисмутаза), туф (коефіцієнт подовження Tu), гроESL (білки теплового шоку) та rpoГени B (b ‐ субодиниці бактеріальної РНК-полімерази) є більш перспективними, але даних недостатньо для остаточних висновків.
В даний час найнадійніша ідентифікація VGS може бути досягнута за допомогою аналізу кількісних окулярів (MLSA) 14. Цей підхід заснований на побудові філогенетичного дерева на об'єднаних послідовностях семи фрагментів ведення домашнього господарства та відображенні клональних зв'язків між невідомими штамами, що досліджуються, та штамами, що зберігаються у відкритих базах даних. http://viridans.emlsa.net/ дозволяє віднести штами стрептококів до видів у межах VGS. Правильно S. pneumoniae ідентифікація також може бути здійснена за допомогою бази даних mlst.net, яка була розроблена головним чином для внутрішньовидового типізації пневмококів 15. На жаль, MLSA є відносно дорогим і трудомістким.
Нещодавно пряме бактеріальне профілювання за допомогою матрично-допоміжної лазерно-десорбційної іонізації – часу польоту мас-спектрометрії (MALDI-TOF MS) було запропоновано як інструмент для швидкої ідентифікації різних бактерій. На жаль, використовуючи базу даних Biotyper 3.0 (Bruker Daltonics, Бремен, Німеччина) Стрептокок mitis/oralis можна помилково визначити як S. pneumoniae, через виняткову схожість їх мас-спектрів 16-19 .
У цьому дослідженні було застосовано ряд математичних класифікаційних алгоритмів для сортування мас-спектрів VGS шляхом відбору набору масових піків, що розрізняють фенотипово та генетично охарактеризовані ізоляти різних видів VGS. З цих класів було сформовано ряд класифікаційних моделей, які порівняно за параметрами чутливості та специфічності. Нарешті, вдалі моделі пройшли сліпе тестування на випадково обраних S. pneumoniae і S. mitis штами.
Методи
Штами
Загалом у дослідження було включено 62 VGS. Тридцять чотири з них були визначені в нашому попередньому дослідженні як S. mitis і три як S. oralis 20. Двадцять п’ять ізолятів були ідентифіковані як S. pneumoniae на основі розчинності жовчі, сприйнятливості до оптохінів та позитивних результатів тесту «Slidex ® pneumo-kit» (bioMerieux ®, Marcy-l'Etoile, Франція). Для S. pneumoniae ізоляти, серотипи визначали з використанням антисироватки, отриманої від Інституту сироватки крові (Staten Serum Institute) (Копенгаген, Данія) відповідно до рекомендацій виробника. Всі ізоляти VGS зберігали при -80 ° C у флаконах CRYOBANK ™ (Копан, Італія).
До аналізу мас-спектрометрії та генетичних досліджень ізоляти пересівали на агарі Колумбія (Oxoid Ltd, Бейсінгсток, Великобританія) з додаванням 5% овечої крові (інкубація протягом ночі при температурі 35 ° C на повітрі з 5% CO2) та фенотиповими тестами (розчинність у жовчі, сприйнятливість до оптохіну та тест `` Slidex ® pneumo-kit '') повторювались. Тест на чутливість до оптохінів проводили із застосуванням стандартних діагностичних оптохінових дисків (Науково-дослідний центр фармакотерапії, Санкт-Петербург, Росія), на повітрі з 5% СО2.
Аналіз генів: схеми MLST та MLSA
Для генетичних маніпуляцій ДНК стрептококів витягували за допомогою набору «ДНК-експрес» (ТОВ «Lytech», Москва, Росія), відповідно до інструкцій виробника. MLST та MLSA виконувались, як описано Enright та Spratt 15 та Bishop та ін. 14 відповідно. Результати аналізували за допомогою баз даних MLST (http://www.mlst.net) та MLSA (http://viridans.emlsa.net/).
Збір даних MALDI TOF
Для аналізу мас-спектрометрії MALDI-TOF дві-три ізольовані колонії свіжих бактеріальних культур (18 год) відбирали стерильною пластиковою петлею об'ємом 1,0 мкл (FL Medical, Торреглія, Італія) і суспендували в 300 мкл чистої води (Fluka, Сент-Луїс, Міссурі, США). Після осадження етанолом (900 мкл) гранулу обробляли сумішшю мурашиної кислоти/ацетонітрилу. Бактеріальні екстракти (1 мкл) були помічені на мішень для зразка MALDI, покриті 1 мкл матриці (насичений розчин альфа-ціано-4-гідрокси коричної кислоти (CHCA) у 50% ацетонітрилі/2,5% TFA) і висушені в повітря. Мас-спектри реєстрували на мас-спектрометрі Microflex MALDI-TOF (Bruker Daltonics), обладнаному лазером N2 337 нм. З кожного ізоляту було отримано не менше чотирьох мас-спектрів із загальною кількістю 250 пострілів на спектр (50 пострілів у кожному з п’яти різних позицій плями). Для візуального контролю спектрів було використано програмне забезпечення FlexAnalysis 2.4 (Bruker Daltonics). Для порівняння та ідентифікації спектрів було використано програмне забезпечення BioTyper 3.0 (Bruker Daltonics).
Обробка даних за допомогою програмного забезпечення ClinProTools
Для розпізнавання пептидних зразків було використано програмне забезпечення ClinProTools 2.1 (Bruker Daltonics). Це програмне забезпечення спочатку пропонувалось як частина системного рішення ClinProt ™ для пошуку біомаркерів у рідинах людського організму (сироватка, плазма, сеча, слина, церебральна спинномозкова рідина тощо), пов’язаних з різними захворюваннями. Це дозволяє вимірювати та візуалізувати різницю пептидів та білків у мас-спектрах різних зразків 21. Операції попередньої обробки спектра, збору піків та обчислення піків проводились із використанням стандартного режиму. Моделі класифікації були створені з використанням алгоритмів генетичного алгоритму (GA), керованої нейронної мережі (SNN) та QuickClassifier (QC).
Для кожної моделі здатність розпізнавання (RC), відносна кількість правильно класифікованих спектрів для даної моделі за умови, що всі випробувані дані раніше використовувались для визначення моделі, та перехресна перевірка (CV), кількісна міра для були розраховані надійність моделі, яка може бути використана для прогнозування поведінки моделі в майбутньому.
Зовнішню перевірку проводили на сліпо відібраних ізолятах; були визначені значення чутливості та специфічності.
Результати
Аналіз MLST та MLSA
MLST проводили на 25 ізолятах S. pneumoniae, 34 з S. mitis і три з S. oralis. Всі S. pneumoniae ізоляти були сприйнятливі до оптохіну; дев'ять з них належать до серотипу 23F, від п'яти до 6B, від п'яти до 19F, від двох до 18, а серотипи 14, 19A, 9L та 35F були представлені по одному ізоляту кожен.
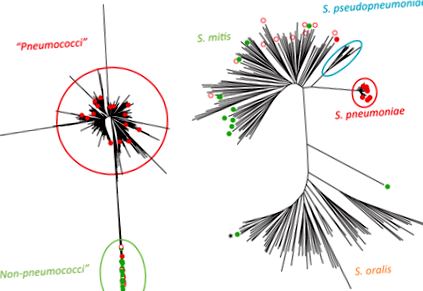
Дендрограма, що зображує клонові зв'язки між ізолятами, представлена на рис. 1 (а). На двох S. oralis ізолятів, він не зміг посилити спі генного локусу, тому ці штами було відкинуто.
Більшість ізолятів групи `` пневмококи '' (24 з 25) потрапляли в кластер пневмококів (рис. 1а, вгорі на малюнку), тоді як ізоляти mitis і oralis утворювали окремий ("непневмококовий") кластер. Для ізолятів кластера «непневмококів» було встановлено, що алелі всіх фрагментів генів принаймні на один або більше відсотків відрізняються від будь-якого відомого для алелю пневмокока. Лише один штам (43 741) групи «пневмококів» несподівано потрапив у скупчення «непневмококів». Цей штам був стабільно сприйнятливий до оптохіну та серотипований як 19F. Потім ми побудували філогенетичне дерево, використовуючи алгоритми ClonalFrame v. 1.1, які дозволяють враховувати можливі рекомбінації в популяціях бактерій; розподіл штамів виявився однаковим (дані не наведені).
Випадково відібрані ізоляти з пневмококової (n = 13) та від непневмококових (n = 22) скупчення, включаючи два S. oralis ізоляти негативні на спі локусу, додатково аналізували за схемою MLSA.
Дендрограма, заснована на об'єднаних послідовностях семи генів ведення домашнього господарства з ізолятів, включених у дослідження, та 244 штамів групи мітису (S. mitis, S. pneumoniae, S. pseudopneumoniae і S. оральний), що зберігається в базі даних http://viridans.emlsa.net/ (Рис. 1b). Усі ізоляти з «пневмококового» кластеру, як визначено аналізом MLST, потрапили в S. pneumoniae кластеру на аналізі MLSA, тоді як 21 із 22 ізолятів із кластеру «непневмококових», включаючи ізолят 43 741, потрапили в S. mitis групи. Це був один ізолят із "непневмококового" скупчення, яке згрупували в S. oralis відділення, разом з двома S. oralis ізоляти, для яких спі локус не вдалося посилити в схемі MLST (вони позначені зірочками на рис. 1b).
Частина ізолятів, визначених MLSA як S. pneumoniae (n = 13) або S. мітит (n = 21) сформував основні групи для генерації класифікаційних моделей. Три штами, віднесені до S. oralis сформували групу ‘oralis’. Інші ізоляти, включені в дослідження (11 S. pneumoniae і 14 S. mitis) сформували допоміжні групи для перевірки моделей.
Збір даних мас-спектрометрії та формування класифікаційної моделі
Близько 120 піків із співвідношенням сигнал/шум більше 5 було виявлено між m/z 2000 і 20000 в мас-спектрах 62 досліджуваних ізолятів.
Усі ізоляти кластера «пневмококів» (див. Рис. 1) були визначені за допомогою програмного забезпечення BioTyper як S. pneumoniae із спектральною оцінкою ≥2,3. Більшість ізолятів кластеру «непневмококових» були ідентифіковані як S. pneumoniae з оцінкою ≥2,0, а деякі ізоляти були визначені як S. pneumoniae або S. oralis, або S. pseudopneumoniae, або S. cristatus, з оцінками не менше 2,0.
Три ізоляти S. oralis скупчення (рис. 2) також були віднесені до S. pneumoniae/S. ораліс/С. pseudopneumoniae/S. cristatus видів.
Далі, мас-спектри, отримані від штамів основної групи, 13 з S. pneumoniae та 21 з S. mitis, були порівняні за допомогою програмного забезпечення ClinProTools 2.1. За допомогою алгоритмів GA, SNN та QC було створено ряд різних моделей класифікації. RC, CV, чутливість та специфічність моделей представлені в таблиці 1.
| GA | 100,00 | 100,00 | 100,0 | 98,6 |
| SNN | 100,00 | 98,65 | 100,0 | 100,0 |
| Контроль якості | 100,00 | 99,46 | 100,0 | 100,0 |
| QC ‐ 3a a Модель, породжена трьома піками (6949, 9876 та 9975 м/з), що надають значну вагу класифікації. | 98,40 | 98,44 | 97,9 | 100,0 |
- a Модель, породжена трьома піками (6949, 9876 та 9975 м/з), що надають значну вагу класифікації.
У моделі GA кількість піків може бути визначена користувачем. Змінюючи цей параметр, ми створили модель, в якій 17 піків давали 100% значення RC та CV (табл. 1). Кількість піків в обох інших моделях, SNN та QC, було виявлено автоматично. У моделі SNN було визначено шість піків, а в моделі QC - п'ять. Значення CV у 100% було досягнуто лише для моделі GA, тоді як в інших моделях воно було трохи менше. Зверніть увагу, що перехресна перевірка є варіантом автоматичної перевірки під час генерації моделі: невелика частина всіх спектрів залишається виключеною при генерації моделей та кластерному аналізі, потім ці спектри класифікуються та визначається кількість правильних та неправильних прогнозів класу. Ця процедура повторюється кілька разів, і для кожного класу накопичуються правильні та неправильні передбачення класу.
Було три піки (6949, 9876 та 9975 м/з), що надавали максимальні статистичні ваги, які були знайдені в кожній створеній моделі. На рис. 2 (а) показаний приклад піку m/z 6949, що суттєво сприяє моделі видової дискримінації: можна помітити, що цей незначний пік виникає майже у всіх S. mitis ізолює мас-спектри і відсутня в S. pneumoniae мас-спектри. Двовимірний розподіл для двох піків (6949 та 9975 м/z) представлений на рис. 2 (b). Відповідно до цієї діаграми, навіть цих двох піків було достатньо для переконливої дискримінації стрептококових видів. Насправді за форсування лише цих трьох піків (6949, 9876 та 9975 м/з) у модель (позначену як QC ‐ 3 у таблиці 1) супроводжувалося лише незначним зниженням значень RC та CV (98,40% та 98,44% відповідно ).
Кожну з моделей оцінювали наосліп, використовуючи 25 ізолятів допоміжної групи (11 пневмококів та 14 S. mitis). Усі три моделі показали майже 100% значення чутливості та специфічності (табл. 1).
Кількість S. oralis ізоляти (n = 3) було недостатньо для створення надійної моделі. Однак короткі обчислення показали, що всі класифікатори мали змогу робити дискримінацію S. pneumoniae від S. oralis ізолює, але ні S. mitis від S. oralis ізоляти.
Обговорення
Точна ідентифікація ізолятів VGS на видовому рівні є важливою як з точки зору практики, так і для розуміння патогенних механізмів конкретного виду. Для стрептококів групи мітису особливо важко визначити види. Численні підходи, що використовують різні фенотипові рисові ознаки, не дають належної чутливості і не можуть вважатися надійними.
Впровадження профілювання бактеріального білка на основі MALDI-TOF значно покращило процес ідентифікації мікроорганізмів у звичайній практиці. Цей підхід заснований на дослідженні складних мас-спектрів пептидів та дрібних білків, які містять унікальні м/з «Підписи» для різних мікроорганізмів через властиві їм зміни маси і дозволили успішно ідентифікувати та розрізнити широкий спектр організмів. Перші спроби дискримінації VGS за допомогою MS MALDI-TOF були багатообіцяючими 16, 19, 22, але згодом стало ясно, що Biotyper 3.0 (Bruker) не в змозі вирішити проблему 17, 18, 23, 24. Зовсім недавно була представлена нова система Vitek MS (bioMérieux), яка показала відносно хороші результати 25 .
- Нова інноваційна послуга Celesio UK надає можливість пацієнтам отримувати рятувальні процедури
- Безкоштовні клініки, що базуються на доходах, приморський Каліфорнія
- Хтось насправді зумів позбутися своєї мами Нетмумів
- DD Fitness Продукти для схуднення та особи, що впливають на Інтернет, які брешуть своїм послідовникам; Donegal Daily
- Фруїтарна пара вважає, що їх дієта допомагає їм залишатися молодими Metro News