Висока інформаційна здатність зберігання даних на основі ДНК із розширеними кодуючими символами з використанням вироджених основ
Предмети
Анотація
Зберігання даних на основі ДНК стало перспективним методом задоволення експоненціально зростаючого попиту на зберігання інформації. Однак практична реалізація зберігання даних на основі ДНК залишається проблемою через високу вартість запису даних за допомогою синтезу ДНК. Тут ми пропонуємо використовувати вироджені бази як кодуючі символи на додаток до A, C, G та T, що збільшує обсяг даних, які можна зберігати на довжину розробленої послідовності ДНК (інформаційна ємність), і зменшуючи кількість ДНК синтез на зберігання одиниці даних. Використовуючи запропонований метод, ми експериментально досягли інформаційної ємності 3,37 біт/символ. Продемонстрована інформаційна спроможність більше ніж удвічі порівняно з найвищою інформаційною спроможністю, яку досягли раніше. Запропонований метод може бути інтегрований із синтетичними технологіями в майбутньому для зниження вартості зберігання даних на основі ДНК на 50%.
Вступ
Тут ми пропонуємо та демонструємо використання вироджених основ (поєднання чотирьох основ ДНК, які можна вставити в будь-які базові ділянки в послідовності) 11 як додаткових символів кодування для перевищення теоретичної межі інформаційної ємності 2,0 біта/символ. Вироджені основи розташовані в послідовності ДНК, коли нуклеотиди змішуються в певному положенні в послідовності ДНК. Наприклад, у послідовності «AWC», «W» позначає комбінацію A і T; таким чином, у пулі молекул існують два типи варіантів нуклеотидів: «AAC» та «ATC». У цій статті, використовуючи одинадцять вироджених основ на додаток до чотирьох символів ДНК, ми експериментально досягаємо інформаційної ємності 3,37 біт/символ у бібліотеці олігонуклеотидів, що складається з сотень копій кожної послідовності. Іншими словами, ми зберігаємо більше даних, використовуючи менше копій кожної послідовності, порівняно з числом молекули, використаною в попередніх дослідженнях. Як результат, ми демонструємо, що довжина ДНК, необхідна для зберігання однакового обсягу даних, зменшилась більш ніж наполовину порівняно з попередніми звітами 3,4,5,6,9,10. Запропонована технологія може бути інтегрована із синтетичними технологіями в майбутньому, щоб зменшити вартість зберігання даних на основі ДНК на 50%.
Результати
Додавання вироджених основ до зберігання даних на основі ДНК
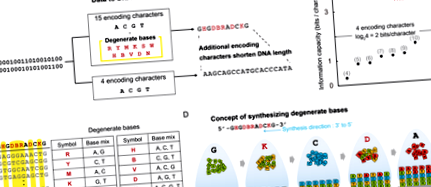
Зберігання даних на основі ДНК з додаванням вироджених основ забезпечує збільшення інформаційної ємності. (A) Двійкові дані кодуються в послідовності ДНК, що містять не тільки 4 традиційні кодуючі символи A, C, G та T, але також 11 додаткових вироджених основ. Довжина закодованої ДНК менше довжини чотирисимвольного методу кодування. (B) Тому обмеження теоретичної інформаційної ємності збільшено з 2 біт/символ до 3,9 біт/символ. Точки на графіку описують значення інформаційної ємності в попередніх дослідженнях, а цифри вказують відповідне посилання. (C.) Вироджена основа, представлена кодуючим символом, описує змішаний пул з більш ніж двох типів нуклеотидів. (D) Вироджені основи можуть утворюватися змішуванням фосфорамідитів ДНК під час синтезу.
Структура та результат декодування платформи зберігання даних на основі ДНК
Структура та результат декодування платформи зберігання даних на основі ДНК. Ми досягли найвищої інформаційної ємності та фізичної щільності зберігання даних на основі ДНК. (A) Проектна структура фрагментів ДНК. (B) Фрагменти ДНК можна аналізувати за допомогою NGS. Після класифікації за адресою вироджені бази можна розшифрувати, вивчивши розподіл символів у тому самому положенні (жовта смужка). (В) Вироджені основи можна визначити за графіком розсіювання співвідношення основ в одному і тому ж положенні. (D) Частота помилок визначених основ ДНК у конкретному середньому охопленні загальних фрагментів. Стандартні відхилення (s.d.) були отримані повторенням випадкової вибірки 10 разів. Смужки помилок представляють s.d. (Е) Підсумок експериментальних результатів. Інформаційна ємність обчислюється на основі вхідної інформації в бітах, поділеної на кількість символів кодування (за винятком кількості сайтів адаптерів). Ми порівняли результати нашої роботи з результатами Ерліха та Зелінських 10, які раніше повідомляли про найвищу інформаційну ємність та фізичну щільність за допомогою об'єднаного синтезу оліго та даних про послідовність з високою пропускною здатністю. Фізична щільність - це відношення кількості байтів, закодованих до ваги бібліотеки ДНК, яка використовується для декодування інформації.
Перевірка та прогнозування вартості запропонованої платформи за допомогою моделювання
0,05 $/100 нт, додаткова примітка) 22 з використанням синтезатора пулу олігонуклеотидів на основі струменевого струменя. Більше того, оскільки вартість секвенування ДНК зменшується швидше, ніж закон Мура, і швидше, ніж синтез ДНК, розрив у цінах між секвенуванням та синтезом збільшиться на замовлення, якщо поточна тенденція збережеться 1,23. Коли застосовується ця вартість, навіть якщо запропонована платформа має надзвичайний випадок покриття NGS у 2000 разів, вартість зчитування даних становитиме менше 5% від вартості запису та менше 0,5%, що буде незначним, через п’ять років (рис. . 3B). Якщо припустити, що синтезатор олігонуклеотидів на основі струменевого принтера встановлений для синтезу виродженої основи, запропонована платформа оцінила зниження вартості зберігання даних на основі ДНК до $ 2052/1 МБ при використанні 15 символів кодування та $ 1795/1 МБ при використанні 21 символу кодування, що є приблизно 50% від попереднього мінімуму $ 3555/1 Мб 10 (Рис. 3B, Додаткова примітка).
Обговорення
У цій демонстрації завдяки використанню вироджених баз інформаційна ємність та фізична щільність були більш ніж подвоєні порівняно з попередньо повідомленими платформами зберігання даних на основі ДНК. Зокрема, із збільшенням інформаційної ємності платформа скорочує довжину ДНК, необхідної для зберігання еквівалентного обсягу даних, і зменшує загальні витрати на зберігання даних наполовину. У наступних дослідженнях фізична щільність буде збільшуватися емпіричним шляхом, а також будуть виконуватися дослідження, що пересувають верхню межу фізичної щільності. Крім того, введений спосіб скорочує час синтезу, якщо є відповідна система синтезу. Наприклад, техніка синтезу олігонуклеотидів на основі колон, яка використовує етапи промивання та зняття захисту, яка збільшується пропорційно довжині синтезованих олігонуклеотидів. Оскільки ми можемо скоротити тривалість синтезу для зберігання однакової кількості даних, час синтезу зменшиться.
Матеріал та методи
Кодування даних до послідовності ДНК
Для першої демонстрації текстовий файл (txt) із описом короткого вступу та списку членів лабораторії, до якої належить відповідний автор, був закодований до ДНК (рис. S1). Для другої демонстрації було закодовано ескіз рукопису Хунміньонгум (рис. S2). Розмір файлу зображення змінено до 692 × 574, а розмір файлу - 135 393 байт. Двійкові дані були витягнуті з файлу та згруповані як довжина фрагмента ДНК. Для другої демонстрації були додані фрагменти надмірності Рід-Соломона. Після цього було додано адресу. Усі цифри були перетворені в ДНК-кодони, як описано в таблицях S1 – S3. Детальніше про дані, що кодують ДНК, описано в Додатковій примітці.
Підготовка та кількісне визначення зразків ДНК
Ампліфікація та секвенування ДНК
Зразки ампліфікували за допомогою qPCR (FAST 7500, Applied Biosystems) та KAPA HiFi Library Amplification Kit. Використовували пробну суміш 10 мкл основної суміші, 6 мкл води для ПЛР, 1 мкл 10 мкМ вихідного матеріалу вперед і назад, 1 мкл розчину для басейну оліго, 20X SYBR Green. Ми дотримувались стандартного теплового протоколу з посібника. Ми перевірили графік ампліфікації за допомогою qPCR. Як тільки графік досяг насичення, ми зупинили машину та очистили відбір проб за допомогою набору для очищення ПЛР (Qiagen). Ми секвенували посилений пул оліго, використовуючи на Miniseq, використовуючи протокол зчитування з парним кінцем 300 циклів.
ДНК для декодування даних
Парне читання необробленого файлу NGS (формат Fastq) було зшито за допомогою PEAR. Після цього зчитування NGS із відповідною довжиною було відфільтровано, а дубльовані зчитування видалено. Дубльовані зчитування були вилучені та зображено послідовність (включаючи вироджену основу). З репрезентуючої послідовності кодон ДНК трансформувався в цифру, дотримуючись Додаткових таблиць S1 – S3. Виправлення помилок за допомогою коду Ріда-Соломона було проведено для другої демонстрації. Детальніше про розшифровку ДНК до даних описано в Додатковій примітці.
Моделювання Монте-Карло
Наявність даних
Набори даних, використані та/або проаналізовані під час поточного дослідження, доступні у відповідного автора на обґрунтований запит.
Список літератури
Жирнов, В., Задеган, Р. М., Сандху, Г. С., Черч, Г. М. і Хьюз, В. Л. Пам'ять нуклеїнових кислот. Нат. Матер. 15, 366–370 (2016).
Clelland, C. T., Risca, V. & Bancroft, C. Приховування повідомлень у мікроточках ДНК. Природа 399, 533–534 (1999).
Bancroft, C., Bowler, T., Bloom, B. & Clelland, C. T. Довготривале зберігання інформації в ДНК. Наука (80-.). 293, 1763c – 1765 (2001).
Голдман, Н. та ін. На шляху до практичного зберігання інформації у синтезованій ДНК з великою ємністю та низьким рівнем обслуговування. Природа 494, 77–80 (2013).
Church, G. M., Gao, Y. & Kosuri, S. Зберігання цифрової інформації наступного покоління в ДНК. Наука 337, 1628 (2012).
Борнхольт, Дж. та ін. Архівна система зберігання даних на основі ДНК - Microsoft. Дослідження. Огляд операційних систем ACM SIGOPS 50, 637–649 (2016).
Блават, М. та ін. Виправлення помилок вперед для зберігання даних ДНК. Procedia Comput. Наук. 80, 1011–1022 (2016).
Органік, Л. та ін. Випадковий доступ у масштабному сховищі даних ДНК. Нат. Біотехнол, https://doi.org/10.1038/nbt.4079 (2018).
Грасс, Р. Н., Геккель, Р., Пудду, М., Паунеску, Д. і Старк, В. Дж. Міцне хімічне збереження цифрової інформації про ДНК в діоксиді кремнію з кодами, що виправляють помилки. Енджу. Хім. Міжнародний Ред. Англ. 54, 2552–5 (2015).
Ерліх, Ю. та Зелінськ, Д. Фонтан ДНК забезпечує надійну та ефективну архітектуру зберігання. Наука (80-.), 950–954 (2017).
Корніш-Боуден, А. Номенклатура неповністю зазначених основ в послідовностях нуклеїнових кислот: рекомендації 1984. Нуклеїнові кислоти Res. 13, 3021–30 (1985).
Beaucage, S. L. & Iyer, R. P. Досягнення в синтезі олігонуклеотидів підходом фосфорамідиту. Тетраедр 48, 2223–2311 (1992).
ЛеПруст, Е. М. та ін. Синтез високоякісних бібліотек довгих (150 метрів) олігонуклеотидів новим контрольованим процесом депуринації. Нуклеїнові кислоти Res. 38, 2522–2540 (2010).
Клірі, М. А. та ін. Виробництво складних бібліотек нуклеїнових кислот з використанням високопаралельних на місці синтез олігонуклеотидів. Нат. Методи 1, 241–248 (2004).
Хьюз, Т. Р. та ін. Експресійне профілювання з використанням мікрочипів, виготовлених струменевим синтезатором олігонуклеотидів. Нат. Біотехнол. 19, 342–347 (2001).
Прикладні біосистеми. Оцінка та виділення синтетичних олігонуклеотидів - Повне керівництво. (1992).
Hecker, K. H. & Rill, R. L. Аналіз помилок хімічно синтезованих полінуклеотидів. Біотехніка 24, 256–60 (1998).
Airaksinen, A. & Hovi, T. Модифіковані основні композиції при вироджених положеннях мутагенного олігонуклеотиду посилюють випадковість у мутагенезі насичення сайтом. Нуклеїнові кислоти Res. 26, 576–581 (1998).
Kosuri, S. & Church, G. M. Масштабний синтез ДНК de novo: технології та застосування. Нат. Методи 11, 499–507 (2014).
Ейрд, Д. та ін. Аналіз та мінімізація упереджень ампліфікації ПЛР у бібліотеках секвенування Illumina. Геном Біол. 12, R18 (2011).
Вільямс, Р. та ін. Ампліфікація складних бібліотек генів за допомогою емульсійної ПЛР. Нат. Методи 3, 545–550 (2006).
Веттерстранд, К. Витрати на секвенування ДНК: дані Програми секвенування геному NHGRI (GSP). Natl. Гул. Геном Res. Ін-т.
Карр, П. А. і Черч, Г. М. Інженерія геномів. Нат. Біотехнол. 27, 1151–1162 (2009).
Чжан, Ю. та ін. Напівсинтетичний організм, який зберігає та отримує підвищену генетичну інформацію. Природа 551, 644–647 (2017).
Подяка
Цю роботу підтримав Центр фінансування досліджень Samsung компанії Samsung Electronics за номером проекту SRFC-IT1601-08.
Інформація про автора
Приналежності
Департамент електротехніки та обчислювальної техніки, Сеульський національний університет, 1, Gwanak-ro, Gwanak-gu, Сеул, 08826, Республіка Корея
Yeongjae Choi, Taehoon Ryu, Hansol Choi, Hansaem Lee, Jaejun Park & Sunghoon Kwon
Міждисциплінарна програма з біоінженерії, Сеульський національний університет, 1, Gwanak-ro, Gwanak-gu, Сеул, 08826, Республіка Корея
Amos C. Lee & Sunghoon Kwon
Департамент електронної техніки, Університет Кен Хі, Донгьон-Даеро, Гіхен-гу, Йонгін-сі, Кьонгі-до, 17104, Республіка Корея
Сук-Хен Сонг, Соджоо Кім, Хієлі Кім і Парк Вук
Інститут підприємницької біоконвергенції, Сеульський національний університет, 1, Gwanak-ro, Gwanak-gu, Сеул, 08826, Республіка Корея
Інститут біомедичних досліджень Національної університетської лікарні Сеула, Національна університетська лікарня Сеула, 101, Daehak-ro Jongno-gu, Сеул, 03080, Республіка Корея
Поточна адреса: Celemics Inc., 131, Gasandigital 1-ro, Geumcheon-gu, Сеул, 08506, Республіка Корея
Таехун Рю та парк Джеджун
Ви також можете шукати цього автора в PubMed Google Scholar
Ви також можете шукати цього автора в PubMed Google Scholar
Ви також можете шукати цього автора в PubMed Google Scholar
Ви також можете шукати цього автора в PubMed Google Scholar
Ви також можете шукати цього автора в PubMed Google Scholar
Ви також можете шукати цього автора в PubMed Google Scholar
Ви також можете шукати цього автора в PubMed Google Scholar
Ви також можете шукати цього автора в PubMed Google Scholar
Ви також можете шукати цього автора в PubMed Google Scholar
Ви також можете шукати цього автора в PubMed Google Scholar
Ви також можете шукати цього автора в PubMed Google Scholar
Внески
Y.C., T.R., W.P. та С.К. ініціювали та розробляли експерименти. Y.C., A.C.L., W.P. та С.К. написав рукопис. Y.C., T.R., A.C.L., H.C., H.L., J.P., S.S., S.K. та Х.К. проводив дослідження, включаючи синтез та аналіз ДНК.
Автори-кореспонденти
Декларації про етику
Конкуруючі інтереси
Y.C., T.R., S.S., S.K., H.K., W.P. та С.К. є винахідниками патентної заявки на спосіб, описаний у цій роботі. Решта авторів не заявляють про конфлікт інтересів.
Додаткова інформація
Примітка видавця: Springer Nature залишається нейтральним щодо юрисдикційних вимог в опублікованих картах та інституційних приналежностей.
- Лабораторія з використанням транзистора для управління сильними струмами навантаження за допомогою Arduino; ITP Фізичні обчислення
- Вплив вибору середовища та піногасника на виробництво та якість моноклональних антитіл з використанням високого рівня
- Чи здорово риба-білянка в Омега-3
- Скутер на говерборді чи є користь для здоров’я від використання говербордів; King Power Boards - Канада; s
- Чи вимагає ваш ресторан законом розміщувати інформацію про харчові продукти MenuTrinfo