Структура метилювання ДНК у жінок із надмірною вагою при дієті з обмеженим енергоспоживанням, доповненою риб’ячим жиром
1 Департамент харчування, харчових наук та фізіології, Університет Наварри, C/Irunlarrea 1, 31008 Памплона, Іспанія
2 Кафедра фізіології та біофізики, Інститут біомедичних наук, Університет Сан-Паулу, проспект професора Лінеу Престес, 1524, 05508-900 Сан-Паулу, Іспанія, Бразилія
3 CIBER Fisiopatología de la Obesidad y la Nutrición (CIBERobn), Instituto de Salud Carlos III, 28029 Мадрид, Іспанія
Анотація
1. Вступ
Отже, метою цього дослідження було перевірити гіпотезу, чи впливає добавка риб'ячого жиру в рамках дієтичного режиму з обмеженим використанням енергії на схему метилювання ДНК генів, які регулюються n-3 Добавки PUFA в PBMC, такі як рецептори жирних кислот CD36 і FFAR3, ферменти FADS1 і PDK4, і поверхневий антиген CD14.
2. Методи
2.1. Дизайн випробувань та учасники
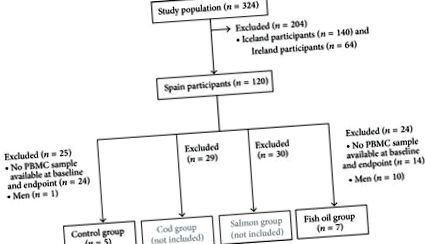
Первинне дослідження базувалося на рандомізованому 8-тижневому дослідницькому дослідженні чотирьох ізокалорійних дієт, призначеного для вивчення специфічних наслідків споживання риби або добавок риб’ячого жиру на втрату ваги у молодих дорослих із надмірною вагою (реєстрація дослідження: ClinicalTrials.gov NCT00315770) [18 ]. У цьому клінічному дослідженні загалом було включено 324 особи з надмірною вагою (140 з Ісландії, 120 з Іспанії та 64 з Ірландії) і випадковим чином розподілені до чотирьох дієтичних груп (капсули контролю, тріски, лосося та риб'ячого жиру) [18]. Детальний опис протоколу, набір та зарахування учасників, критерії включення та виключення описані в інших місцях [18, 19]. Коротко кажучи, початковими критеріями включення були індекс маси тіла (ІМТ) 27,5–32,5 кг/м 2, вік від 20 до 40 років та обхват талії ≥94 см та ≥80 см для чоловіків та жінок відповідно. Критеріями виключення були зміни ваги через дієту для схуднення протягом 3 місяців до початку дослідження, використання добавок, що містять n-3 жирні кислоти, кальцій або вітамін D протягом останніх 3 місяців, медикаментозне лікування цукрового діабету, гіпертонії або гіперліпідемії, а також вагітність або лактація жінок.
). Щоб зменшити мінливість, суб'єкти, що належать до груп тріски та лосося, не аналізувались, оскільки якби в цих групах спостерігалися зміни метилювання ДНК, було б неможливо знайти відповідальних агентів через великі відмінності в макроелементах та мікроелементах між обома видами риб. Дослідження було схвалено Етичним комітетом Університету Наварри та відповідало рекомендаціям Гельсінкі. Усі учасники дали свою письмову згоду після того, як були проінформовані про природу мети та можливі ризики дослідження. Це дослідження було проведено в Університеті Наварри в Памплоні, Іспанія, і набір проводився протягом 2004 та 2005 років.
2.2. Дієтичне втручання
2.3. Антропометричні та метаболічні вимірювання
Вимірювання антропометричних параметрів та відбір зразків крові проводили на початковому рівні та після періоду добавки (кінцевої точки), як описано раніше [24]. Рівні глюкози, загального холестерину та триацилгліцеринів у плазмі крові вимірювали за допомогою специфічних колориметричних аналізів (Horiba ABX Diagnostics, Монпельє, Франція) з використанням автоматичної системи (COBAS MIRA, Рош, Базель, Швейцарія), тоді як рівні інсуліну в крові визначали методом ІФА (Mercodia)., AB, Уппсала, Швеція). Індекс оцінки моделі гомеостазу (HOMA-IR) розраховували згідно з Matthews et al. [25] з метою оцінки резистентності до інсуліну.
2.4. Виділення PBMC, вилучення ДНК та перетворення бісульфіту
PBMC виділяли диференціальним центрифугуванням із застосуванням Polymorphprep (Axis Shield PoC AS, Осло, Норвегія), як було описано раніше [26], і зберігали при -80 ° C. ДНК екстрагували за допомогою набору AllPrep DNA/RNA Mini Kit (Qiagen, Hilden, Німеччина). Концентрацію ДНК визначали кількісно за допомогою набору для аналізу dsDNA Quant-iT PicoGreen (Invitrogen, Карлсбад, Каліфорнія, США) та геномної ДНК (2 μg) було перетворено бісульфіт за допомогою набору EpiTect Bisulfite Kit (Qiagen, Валенсія, Каліфорнія, США).
2.5. Профіль метилювання визначається за допомогою мас-спектрометрії MALDI-TOF
Профілі метилювання ДНК п’яти генів аналізували в PBMC: CD36, CD14, FADS1, PDK4, і FFAR3. Вивчені геномні послідовності наведені в таблиці 1S у додаткових матеріалах, доступних в Інтернеті за адресою http://dx.doi.org/10.1155/2014/675021.
По-перше, регіон, що охоплює 3 кб (2000 пар базових вище за течією до 1000 базових пар нижче за течією від стартового місця транскрипції), був обраний з банку генів (http://www.ncbi.nlm.nih.gov/gene) для кожного гена. Острови CpG були ідентифіковані за допомогою програмного забезпечення MethPrimer (http://www.urogene.org/methprimer/) [27]. Тоді як три гени показали CpG-острови (CD14, PDK4, і FADS1), ні FFAR3 ні CD36 показали ці багаті на CpG динуклеотиди регіони. Прогнозовані сайти зв'язування факторів транскрипції були ідентифіковані за допомогою програмного забезпечення AliBaba2 (http://www.gene-regulation.com/pub/programs/alibaba2/index.html) з гомологією 75%. Потім для кожного гена було вибрано область від 300 до 500 п.н. на острові CpG та/або багату передбачуваними факторами транскрипції (табл. 1). Профіль метилювання цих генів був визначений за допомогою технології MassARRAY EpiTyper Sequenom (Sequenom, Сан-Дієго, Каліфорнія, США), яка спирається на специфічне для розщеплення підставу з подальшою масовою спектрометрією MALDI-TOF, як описано раніше [28]. Використані праймери наведені в таблиці 1. Значення метилювання ДНК деяких сайтів CpG не можна виміряти незалежно. Це випадок із сусідніми сайтами CpG та сайтами CpG, включеними до подібних хімічних фрагментів після ферментативного розпаду. Отже, вони були об’єднані в групи та розглядались як незалежний сайт CpG при аналізі.
2.6. Статистичний аналіз
Дані представлені як середнє значення ± SD і були проаналізовані за допомогою SPSS 15.0 для Windows (SPSS, Чикаго, Іллінойс, США). Загальна лінійна модель із повторними заходами була проведена для оцінки впливу пероральних добавок (контролю або риб’ячого жиру) на метилювання ДНК CD36, FFAR3, CD14, PDK4, і FADS1 через лікування схуднення (вихідна та кінцева точки). Для сайту CpG +477 з гена CD36 ми припустили, що різниця у початковій масі тіла може вплинути на статистичний аналіз. З метою контролю впливу маси тіла проводили статистичний аналіз з базовою масою тіла як ковариабельной. Рівень значущості був встановлений на рівні
3. Результати
3.1. Профіль вихідного метилювання ДНК
Набір проводився протягом 2004 та 2005 рр. Загалом для цього дослідження було відібрано 12 жінок (рис. 1). Різні закономірності метилювання були виявлені в генах, що вивчались у PBMC (рис. 2). Низькі рівні метилювання (менше 25% [29]) спостерігалися в генах, що кодують поверхневу молекулу CD14 і метаболічні ферменти PDK4 і FADS1, хоча одна CpG PDK4 (-229-222) був високо метильованим (понад 75% [29]). CD36 досліджуваний регіон був високо метильованим, тоді як FFAR3 показав дві різні області: сильно метильовану область, близьку до місця початку транскрипції (сайти CpG -18, +33 та +77), та область з низьким метилюванням між CpG -53 і -202.
3.2. Лікування схуднення та вплив добавок риб’ячого жиру на профіль метилювання ДНК
Обидві експериментальні групи (контрольна група та риб'ячий жир) реагували аналогічно на лікування схуднення (табл. 2), демонструючи подібне зниження маси тіла, ІМТ та рівня лептину в сироватці крові (), але не впливаючи на інші метаболічні параметри.
Втрата ваги, спричинена гіпокалорійною дієтою, вплинула на метилювання ДНК у CD36 гена, зменшуючи відсоток метилювання CpG +477 (рис. 3 (а)). Цей CpG лежить у передбачуваному місці зв'язування факторів транскрипції CREB та CRE-BP1 (Рисунок 2 (а), CpG +477). Однак добавки риб'ячого жиру не змінили схему метилювання ДНК CD36 гена зі зменшенням на 11,8% метилювання CpG +477 у контрольній групі та 7,3% у групі риб'ячого жиру. З метою контролю впливу маси тіла статус метилювання було відрегульовано до базової маси тіла. У цьому випадку метилювання CpG +477 було нижчим () у групі риб'ячого жиру (6,7%), ніж у контрольній групі (12,7%).
). Метилювання ДНК визначали за зразками базової лінії та кінцевої точки за допомогою MassARRAY EpiTyper у трьох примірниках. Загальна лінійна модель з повторними вимірами була використана для оцінки впливу пероральних добавок на метилювання ДНК (між випробовуваними) для лікування схуднення (у випробовуваних).
, різниці внаслідок втрати ваги (вихідний рівень проти кінцевої точки);
Обмеження енергії також вплинуло на метилювання ДНК у Росії CD14 (Малюнок 3 (c)), але лише з дуже незначним збільшенням на 0,3% та 0,6% при CpGs + 765 + 773 у контрольній та риб'ячому жирах відповідно. Ці CpG включені в передбачуваний сайт зв'язування для факторів транскрипції USF та Sp1 (рис. 2 (c)).
Статистично значущі (хоча і незначні) зміни також були спричинені зниженою вагою гіпокалорійної дієти у промоторах PDK4 і FADS1 гени (CpGs -254 та -25-22-20, відповідно, на малюнках 3 (d) та 3 (e)). Крім того, єдині CpG, які продемонстрували значні відмінності у метилюванні між контролем та групами риб’ячого жиру, є в цих генах. Таким чином, відсоток метилювання PDK4 Сайти CpG −222 та −50 та FADS1 CpG −25−22−20 зменшувались у контрольній групі та збільшувались з риб’ячим жиром, тоді як зменшення спостерігалось у PDK4 CpG -254 у контролі був більшим, ніж у групі риб'ячого жиру. Цікаво, PDK4 CpGs -254 та -50 лежать у передбачуваних сайтах зв'язування для фактора транскрипції Sp1 (рис.2 (d)). Тим не менше, хоча статистично значущі, ці відмінності були настільки незначними, що вони, ймовірно, не впливають на експресію генів.
4. Обговорення
Хоча є вагомі докази модулюючого впливу n-3 PUFA щодо експресії генів у кількох типах клітин у людини, невідомо, чи могли вони впливати на експресію генів, змінюючи епігенетичний механізм метилювання ДНК. Тут ми намагалися визначити профілі метилювання CD36, FFAR3, CD14, PDK4, і FADS1 у PBMC та чи впливає це метилювання на дієтичне втручання для схуднення (обмеження енергії) з добавками риб’ячого жиру чи без нього у молодих жінок із надмірною вагою. Як і слід було очікувати, фенотипічні зміни внаслідок обмеження енергії призвели до зменшення маси тіла. У людини дієти з обмеженим енергією модифікують метилювання ДНК у PBMC [28]. Наші результати підтверджують ці висновки, припускаючи, що лікування схуднення спричиняє незначні зміни у профілі метилювання конкретних генів з клітин крові (тобто., CD36, CD14, PDK4, і FADS1).
- Щоденник зайвої ваги - Отримайте безкоштовний дієтичний блог на
- Епідуральна настільки ж ефективна для жінок із зайвою вагою
- Ефекти гідрохлориду бетагістину у жінок із зайвою вагою - Повний текст
- Чи надмірна вага змушує вас виглядати молодшою (втрата ваги, втрата, втрата) - дієта та втрата ваги
- Вправа перед допоміжним заплідненням у жінок із надмірною вагою та ожирінням (FertilEX)