Послідовність геному жирафа відкриває підказки про його унікальну морфологію та фізіологію
Предмети
Анотація
Невідоме походження імпозантного росту жирафа та пов'язаних із цим серцево-судинних адаптацій. Окапі, якому не вистачає цих унікальних особливостей, є найближчим родичем жирафа і забезпечує корисне порівняння для виявлення генетичних змін, що лежать в основі довгою шиї та серцево-судинної системи жирафа. Геноми жирафа та окапі були послідовно розподілені, і шляхом порівняльного аналізу були виявлені гени та шляхи, які демонструють унікальні генетичні зміни та, ймовірно, сприяють унікальним особливостям жирафа. Деякі з цих генів знаходяться в сигнальних шляхах HOX, NOTCH та FGF, які регулюють розвиток скелета та серцево-судинної системи, що дозволяє припустити, що ріст жирафа та серцево-судинні адаптації еволюціонували паралельно через зміни в невеликій кількості генів. Гени метаболізму мітохондрій та летючих жирних кислот також еволюційно розходяться у жирафа і можуть бути пов'язані з його незвичним харчуванням, яке включає токсичні рослини. Несподівано відбулися суттєві еволюційні зміни у жирафа та окапі у відновленні дволанцюгових розривів та функціях центросом.
Вступ
Окапі (Okapia johnstoni), найближчий родич жирафа і єдиний інший член, що дійшов до нас Жирафи сім'я, надає корисне порівняння, оскільки воно не поділяє цих унікальних атрибутів, що спостерігаються у жирафа 13. Виявлено дев’ять підвидів жирафа, які можна відрізнити за кольором і малюнком шерсті, і репродуктивно були виділені до 2 міль (посилання 14, 15). Два підвиди жирафів майже вимерли, і загалом кількість жирафів зменшилась на 40% з 2000 року через браконьєрство та втрату середовища проживання 16. Оскільки всі підвиди жирафів мають унікальну анатомо-фізіологічну адаптацію роду жирафів, вони забезпечують важливу перехресну перевірку унікальних закономірностей генетичних варіацій.
Тут ми здійснили секвенування геномів масаїського жирафа та окапі, а шляхом порівняльного аналізу з іншими ссавцями евтерій було виявлено 70 генів, які виявляють множинні ознаки адаптації (MSA) у жирафа. Деякі з цих генів кодують добре відомі регулятори розвитку скелета, серцево-судинної та нервової системи, і, ймовірно, сприятимуть унікальним характеристикам жирафа.
Результати
Секвенування геному та de novo складання
Порівняльний аналіз геному
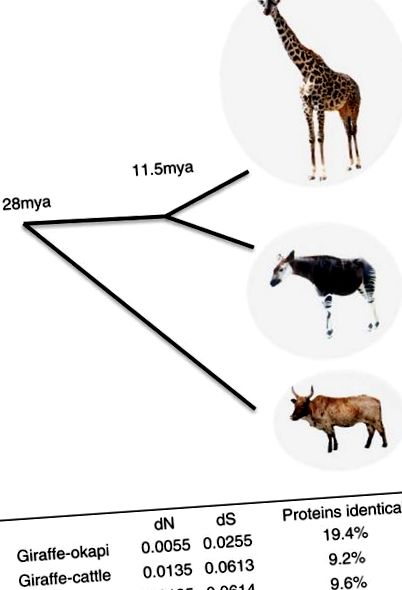
Щоб виявити зміни, які потенційно лежать в основі цих унікальних морфологічних та фізіологічних адаптацій, ми проаналізували кодуючі послідовності ортологічних генів у жирафа, окапі та великої рогатої худоби. Гени жирафа та окапі в цілому дуже схожі - 19,4% білків однакові (рис. 1). Гени жирафа та окапі однаково віддалено пов'язані з великою рогатою худобою, що припускає, що унікальні характеристики жирафа не зумовлені загальним швидшим темпом еволюції. Розбіжність жирафа та окапі, заснована на відносній швидкості заміщення синонімів, від загального предка, за оцінками, становить 11,5 мільйона (рис. 1), що значно менше, ніж попередня оцінка у 16 мільйонів (посилання 19, 20), яка була на основі порівняння послідовностей мітохондріальних ДНК.
Використовуючи середні парні синонімічні розбіжності заміщення (dS) між жирафом, окапі та великою рогатою худобою, відкалібровані загальним предком пекорана (27,6 мільйона), оцінка дивергенції жирафа та окапі від загального предка становить 11,5 міль. Зображення Окапі, адаптоване за фотографією Рауля654.
Адаптивна еволюція жирафа
Було виявлено сімдесят генів, які демонстрували MSA на основі розбіжності амінокислотних послідовностей, оцінених шляхом приєднання сусіднього філогенетичного аналізу ортологічних білків ссавців, збагачення несинонімічних замін, унікальних амінокислотних замін на місцях, які інакше фіксуються у ссавців, замін, які, як передбачається, можуть спричинити функціональні зміни Polyphen2 аналіз та заміни при позитивному відборі. Кластерний аналіз проводили на наборі 70 генів MSA жирафа на основі біологічного процесу GO з використанням Cytoscape 3.0 (посилання 68).
Еволюція регуляторів скелетного росту та диференціації
Giraffe FOLR1 демонструє надзвичайно вагомі докази адаптивної еволюції, включаючи шість позитивно відібраних амінокислотних замін, два з яких, як передбачається, спричинять значну зміну функції (Рис. 3b). FOLR1 мутації є ембріонально летальними у мишей 28 і викликають гіпомієлінізацію та неврологічні дефекти у людей 29. На додаток до своєї ролі у клітинному переносі фолатів, FOLR1 інтерналізується, переробляється та транспортується до ядра, де він регулює компоненти FGF та NOTCH-шляхів 30. Ці зміни в жирафі FOLR1 можуть діяти спільно з подібними змінами в FGFRL1 і JAG1, компоненти FGF та NOTCH, відповідно, для формування основних адаптацій розвитку.
Еволюція серцево-судинних та метаболічних генів
Серцево-судинна система жирафа пристосована для регулювання артеріального тиску на висоті 6 м та для підтримки серцево-судинного гомеостазу, пов'язаного зі швидкими змінами відносного положення мозку до серця. Артеріальний тиск жирафа в 2,5 рази вище, ніж у людини, лівий шлуночок серця збільшений, а стінки судин нижніх кінцівок сильно потовщені 1,31. Жираф демонструє докази адаптивного розвитку восьми генів, які регулюють кров'яний тиск або серцево-судинну функцію, включаючи два основних адренергічні рецептори α1 і β-2, уротензин-2b та ангіотензинперетворюючий фермент (Додаткова таблиця 4). BORG1 та RCAN3, які сильно експресуються в серці та мають важливу функцію, пов'язану з формою клітин та скороченням серцевого м'яза, відповідно, також істотно розходяться у жирафа 32,33. Спостережувані відмінні зміни в цих генах можуть дати підказки щодо еволюційного походження високого кров'яного тиску жирафа, збільшення серцевого викиду та модифікованої судинної системи.
Підвищений зріст жирафа дозволяє йому харчуватися листям акації та насінниками, які є дуже поживними, але містять токсичні алкалоїди. Як і у інших жуйних тварин, мікроби кишечника жирафа ферментують рослини для утворення летких жирних кислот, які транспортуються через епітелій кишечника і служать основним джерелом енергії 34,35. Серед генів MSA у жирафа є ті, хто бере участь у катаболізмі летких жирних кислот, таких як бутират (MCT1, ACSM3 та ACADS) або окисне фосфорилювання, що генерує АТФ (NDUB2 та SDHB) (рис. 3в). Крім того, ці білки необхідні для транспортування лактату та метаболізму, що особливо важливо для серцево-судинних функцій 36 .
Еволюційні зміни в генах відновлення ДНК та хромосом
Обговорення
Розвиваючі та фізіологічні регуляторні гени у жирафа, які проявляють адаптивну еволюцію, збагачуються скелетними, серцево-судинними та нервовими функціями. Перераховані гени MSA, які, як відомо, не пов’язані з регуляцією скелетного, серцево-судинного або нервового розвитку (праворуч).
Серед 70 генів, що демонструють MSA у жирафа, FGFRL1 є найсильнішим кандидатом для безпосереднього впливу на унікальний ріст осьово-апендикулярного скелета та серцево-судинної системи. Відомо, що FGFRL1 має важливе значення для нормального розвитку скелета та серцево-судинної системи у людей та мишей 25,26,27, а шлях FGF регулює розмір соміту 51. Інші гени необхідні для обмеження диференціального росту шийних хребців та ніг, і гомеотичні гени, які визначають ідентичність різних областей тіла, ймовірно, відіграють цю роль. Ми виявили три гени homeobox -HOXB3, CDX4 і NOTO—Котрий демонструє значні зміни у жирафа порівняно з іншими ссавцями. Поява методів редагування генів забезпечує засіб перевірки цих гіпотез шляхом введення унікальних амінокислотних замін, які спостерігаються у жирафа, в гомологічні гени модельних організмів та визначення функціональних наслідків. Серед ссавців у жирафа є найскладніші фізіологічні та структурні проблеми, обумовлені висотою його висоти. Рішення цих проблем, зокрема пов'язані з турбованою системою кровообігу, можуть бути корисними для лікування серцево-судинних захворювань та гіпертонії у людей.
Методи
Секвенування геному
Набір для підготовки бібліотек без ПЛР для ілюмінації TruSeq ДНК був використаний для побудови парних кінцевих бібліотек за зразками печінки двох самок жирафа Масаї (G.c. тіппельскірчі) з MA1 у Кенії та зоопарку Нешвіля (NZOO), а також один фекалієвий самець окапі (О. Джонстоні) з холдингу White Oak. Бібліотеки були підготовлені відповідно до протоколу виробника з використанням 2 мкг вхідних даних та робочого процесу розміром вставки 550 bp. Набір для підготовки зразків пари Nextera Mate Pair був використаний для побудови бібліотек парних пар із тих самих трьох зразків, використовуючи протокол виробника «Gel Plus» із вибором розміру 4–8 кб. Бібліотеки були послідовно розподілені на Illumina HiSeq 2500 в режимі швидкого запуску із використанням парного кінця послідовності 2 × 150 bp. Усі бібліотеки були підготовлені та послідовно розподілені в штаті Penn State Genomics Core Facility в Університетському парку, Пенсильванія. Цільове секвенування конкретних генів у Ротшильда (G.c. rothschildi) і сітчасті (G.c. retulata) жираф використовував геномну ДНК, яку ми виділили з первинних культур клітин фібробластів, отриманих від доктора Олівера Райдера з Інституту досліджень природокористування в зоопарку Сан-Дієго.
Контроль якості та охоплення геному
Міжвидові варіанти нуклеотидів ідентифікували наступним чином. Послідовності, які вирівнювались до еталонного геному, як описано вище, були відсортовані за початковою позицією їх вирівнювання до еталонного геному. Потім вони були зібрані за допомогою довідкового підходу 52, вимагаючи принаймні 2-кратного та максимум 80-кратного охоплення регіону, який слід розглядати для складання. Послідовності з зразків окапі були вирівняні до консенсусної послідовності жирафа, використовуючи BWA 53 версії 0.5.9 із аргументами за замовчуванням, а відмінності між жирафом та окапі були потім визначені за допомогою SAMtools 54 версії 0.1.19 із аргументами за замовчуванням та командою mpileup. Внутрішні сценарії (доступні за запитом) використовувались для визначення положення варіантів щодо посилальної послідовності (корови чи собаки).
Читання відкидали, якщо вищезазначений процес виявляв докази недостатньої якості читання або нестабільності геномної області, використовуючи три критерії. По-перше, зчитування вимагали найкращого вирівнювання до еталонного вузла з принаймні на 3% більше однакових нуклеотидів, ніж друге найкраще вирівнювання. По-друге, посилальні контиги ігнорувались, якщо глибина охоплення була занадто великою або занадто низькою згідно зі статистикою Лендера – Вотермена. По-третє, регіони з надзвичайно високим передбачуваним рівнем міжвидових відмінностей ігнорувались, щоб зменшити вплив дублювання та регіони низької складності. Середня глибина охоплення зчитування для нуклеотидних відмінностей, виявлених за допомогою еталонного вузла собаки та застосованих у подальших аналізах, становила 20,0 для жирафа з MA1, 21,6 для жирафа в зоопарку Нешвілла (NZOO) та 16,8 для окапі.
Приблизно 300 генам, які демонстрували відносно високі співвідношення dN/dS у жирафа порівняно з коровою та окапі, не вистачало повного охоплення щодо великої рогатої худоби чи інших ортологів інших ссавців. У більшості випадків неповне охоплення цих генів було пов’язане з тим, що еталонна модель гена великої рогатої худоби, яка була використана, була неповною щодо інших ссавців. Для заповнення анотації цих генів були ідентифіковані ешафоти жирафа та окапі, що містять ці гени. Відповідні ліси були проаналізовані програмою анотацій Genewise 55 з використанням повних еталонних послідовностей кодування великої рогатої худоби або людини. Були використані довідкові стенограми Ensembl з найвищим ступенем достовірності та інформації (TSL: 1, GENECODE basic, APPRIS P1).
De novo складання
По-перше, адаптери TruSeq з даних пар пари були видалені за допомогою параметрів Nesoni за замовчуванням (v0.115) (https://github.com/Victorian-Bioinformatics-Consortium/nesoni). Потім було виконано KmerGenie (v1.6269) 56 із параметрами за замовчуванням для обох наборів даних, щоб визначити найкращі розміри k-mer для складання. Ліси були зібрані за допомогою SOAPdenovo2 (v2.04) 57, встановивши розмір k-mer на 91 для набору даних жирафа та 81 для набору даних okapi, і дозволивши повторне роздільну здатність (параметр -R). Нарешті, прогалини в лісах були заповнені за допомогою GapCloser (v1.12) із параметрами за замовчуванням.
Ті самі зчитування парних кінцівок та пар, що були використані для складання, були відображені назад до збірки жирафа та окапі. Програма BWA-MEM виконувалась із параметрами за замовчуванням, а статистичні дані витягувались за допомогою інструменту «статистика samtools». Примітно, що відсоток правильно відображених пар подружжя був нижчим, ніж для парних кінців, оскільки більший проміжок пари подружнього партнера робить більш вірогідним картографування на різних лісах.
Вирівнювання та генетичні дерева
Перш ніж вирівнювати послідовності, tblastn запускали на кожній послідовності щодо відповідної послідовності RefSeq коров'ячого білка (завантажено з Ensembl). Це забезпечило корекцію для змінних кадрів, оскільки було зазначено, що деякі послідовності мають якість чернетки і можуть мати деякі помилки послідовності. Послідовності вирівнювали за допомогою випуску MUSCLE 3.8 (посилання 58), а філогенетичні дерева будували за допомогою PhyML версії 3.0 (посилання 59). PhyML використовує вірогідний алгоритм пошуку дерева, щоб знайти оптимальний філогенез. Завантаження (n= 100) було використано для перевірки міцності отриманих філогеній.
Аналіз позитивного відбору
Оцінка замін нуклеотидів та амінокислот
Додаткова інформація
Коди приєднання: Дані послідовності для G. camelopardalis tippelskirchi (MA1 та NZOO) та О. Джонстоні (WOAK) були депоновані в архіві короткого читання під номером проекту SRP071593 (BioProject PRJNA313910) та кодами приєднання NZOO: SRX1624609 та MA1: SRX1624612. Проект "Дробовик цілого геному" G. camelopardalis tippelskirchi (MA1) було депоновано в DDBJ/ENA/GenBank під вступом LVKQ00000000, а версія, описана в цьому документі, є версією LVCL01000000. Проект "Дробовик цілого геному" О. Джонстоні (WOAK) було депоновано в DDBJ/ENA/GenBank під ліцензією LVCL00000000, а версія, описана в цьому документі, є версією LVCL01000000.
Як цитувати цю статтю: Агаба, М. та ін. Послідовність геному жирафа відкриває підказки про його унікальну морфологію та фізіологію. Нат. Комун. 7: 11519 doi: 10.1038/ncomms11519 (2016).
- Інформаційний бюлетень про жирафи Блог Nature PBS
- ЕКСКЛЮЗИВ "Будучи Мері Джейн" Зірка Ворон Гудвін НЕЙТЕРЗУЄ Етикетка Пишних Дівчат
- Вправа для схуднення - Фізіологія вправ Healthsmart
- Шеріл Коул розкриває свої поради щодо вправ та дієти ближче
- Дом Сіблі виявляє втрату ваги майже на два камені через відчуття; сором'язливий; Спортивний крот