Кількісний метод ПЛР у реальному часі, специфічний для виявлення та кількісного визначення першої комерціалізованої рослини, відредагованої геномом
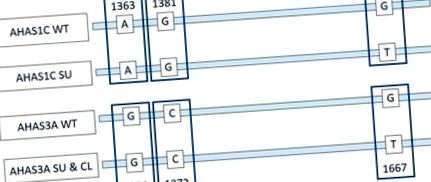
Взаємозв'язок між AHAS1C та AHAS3A та положеннями ключової мутації. Представлення кодуючих ланцюгів гена AHAS1C та гена AHAS3A у дикому типі (WT), геном, відредагований для толерантності до сульфонілсечовини та імідазолінону (SU) та ріпаку Clearfield (CL). Ці області генів AHAS1C та AHAS3A ідентичні, за винятком зазначених основ. Відмінності в нумерації двох генів зумовлені різницею послідовностей вище за течією зображеної області. Два SNV в 1363/1354 і в 1381/1372 відрізняють AHAS1C від AHAS3A. SNV 1676/1667 відрізняє AHAS1C SU, AHAS3A SU та AHAS3A CL від AHAS1C WT та AHAS3A WT. Детальніше про структуру сімейства генів AHAS див. У цитованих у цьому розділі статтях.
Частина секвенування Сангера сегмента гена AHAS1C, що охоплює місце редагування геному, в положенні 1676, виділена синім кольором. Ця основа - гомозиготний G (дикий тип) у сорту каноли Clearfield 5545 CL, гомозиготний T (мутант) у сорту каноли SU 40K та гетерозиготний G/T у сортів ріпаку SU C5507 та C1511. Секвенувана область розповсюджена від специфічних для AHAS1C SNV в положеннях 1363 та 1381 до положення 1704, перевіряючи місце редагування геному в положенні 1676. Місце розташування праймера, що використовується для секвенування, показано на малюнку 3 .
Послідовність частини екзону 2 гена AHAS1C каноли SU, що показує вирівнювання праймерів та зонда для амплікону 334 bp. Прямий праймер та зонд відповідають цільовій послідовності, тоді як зворотний праймер доповнює цільову послідовність. Зірка прямого праймера вказує на G, який є характерним для гена AHAS 1C, відрізняючи його від гена AHAS 3A, який має C на цьому місці. Зірка на зворотному грунтовці вказує на єдиний SNV, що забезпечує толерантність до гербіцидів AHAS 1C SU та AHAS 3A SU та CL. Праймер для секвенування - це олігонуклеотид, який використовується як праймер для секвенування Сангера, представлений на малюнку 2 .
Кількісні криві ампліфікації ПЛР для результатів, наведених у таблиці 2, з праймерами, специфічними для гена CruA (панель A), або з праймерами, специфічними для гена SU ріпаку AHAS1C-SU (панель B). На панелі B ампліфікація відбулася лише у сортів ріпаку SU 40K, C1511 та C5507, тоді як на панелі A посилення також відбулася у сортів ріпаку Clearfield.
Кількісні криві ампліфікації ПЛР для результатів, наведених у таблиці 3. Панель A, ампліфікація за допомогою методу qPCR, специфічного для ендогенного еталонного гена ріпаку (CruA); Панель B, посилення за допомогою методу qPCR, специфічного для SNV AHAS1C-SU каноли SU. Позитивним контролем є ДНК із події каноли SU 40K при 300 (a), 30 (b) та 3 (c) нг 40K ДНК за реакцію.
Типові криві ампліфікації для результатів, наведених у таблиці 3 ДНК сорту каноли сорту 40K, змішаної при п’яти концентраціях із сортом ДНК каноли Clearfield 5545 CL.
Анотація
Взаємозв'язок між AHAS1C та AHAS3A та положеннями ключової мутації. Представлення кодуючих ланцюгів гена AHAS1C та гена AHAS3A у дикому типі (WT), геном, відредагований для толерантності до сульфонілсечовини та імідазолінону (SU) та ріпаку Clearfield (CL). Ці області генів AHAS1C та AHAS3A ідентичні, за винятком зазначених основ. Відмінності в нумерації двох генів зумовлені різницею послідовностей вище за течією зображеної області. Два SNV в 1363/1354 і в 1381/1372 відрізняють AHAS1C від AHAS3A. SNV 1676/1667 відрізняє AHAS1C SU, AHAS3A SU та AHAS3A CL від AHAS1C WT та AHAS3A WT. Детальніше про структуру сімейства генів AHAS див. У цитованих у цьому розділі статтях.
Частина секвенування Сангера сегмента гена AHAS1C, що охоплює місце редагування геному, в положенні 1676, виділена синім кольором. Ця основа - гомозиготний G (дикий тип) у сорту каноли Clearfield 5545 CL, гомозиготний T (мутант) у сорту ріпаку SU 40K та G/T гетерозиготний у сортів ріпаку SU C5507 та C1511. Секвенувана область розповсюджена від специфічних для AHAS1C SNV в положеннях 1363 та 1381 до положення 1704, перевіряючи місце редагування геному в положенні 1676. Місце розташування праймера, що використовується для секвенування, показано на малюнку 3 .
Послідовність частини екзону 2 гена AHAS1C каноли SU, що показує вирівнювання праймерів та зонда для амплікону 334 bp. Прямий праймер та зонд відповідають цільовій послідовності, тоді як зворотний праймер доповнює цільову послідовність. Зірка прямого праймера вказує на G, який є характерним для гена AHAS 1C, відрізняючи його від гена AHAS 3A, який має C на цьому місці. Зірка на зворотному грунтовці вказує на єдиний SNV, що забезпечує толерантність до гербіцидів AHAS 1C SU та AHAS 3A SU та CL. Праймер для секвенування - це олігонуклеотид, який використовується як праймер для секвенування Сангера, представлений на малюнку 2 .
Кількісні криві ампліфікації ПЛР для результатів, наведених у таблиці 2, з праймерами, специфічними для гена CruA (панель A), або з праймерами, специфічними для гена SU canola AHAS1C-SU (панель B). На панелі B ампліфікація відбулася лише у сортів ріпаку SU 40K, C1511 та C5507, тоді як на панелі A посилення також відбулася у сортів ріпаку Clearfield.
Кількісні криві ампліфікації ПЛР для результатів, наведених у таблиці 3. Панель A, ампліфікація за допомогою методу qPCR, специфічного для ендогенного еталонного гена ріпаку (CruA); Панель B, посилення за допомогою методу qPCR, специфічного для SNV AHAS1C-SU каноли SU. Позитивним контролем є ДНК із події каноли SU 40K при 300 (a), 30 (b) та 3 (c) нг 40K ДНК за реакцію.
Типові криві ампліфікації для результатів, наведених у таблиці 3 ДНК сорту каноли сорту 40K, змішаної при п’яти концентраціях із сортом ДНК каноли Clearfield 5545 CL.
- Безкоштовне повнотекстове опромінення ультрафіолетовим випромінюванням прокатного філе шинки, інокульованої ієрсінією
- Повна повнотекстова бузина (Sambucus nigra L
- Генетично модифіковані продукти Шкідливі чи корисні есе - Безкоштовні аргументовані есе для студентів
- Гени Безкоштовний повнотекстовий одомашнений кляп Ген дрозофіли LTR ретротранспозони задіяний у
- Безкоштовне повнотекстове розуміння системи «Вічна мерзлота-гідрат» та пов'язаного з нею метану