Еволюція та класифікація систем CRISPR – Cas
Предмети
Анотація
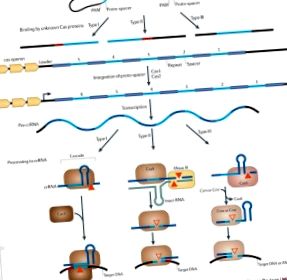
Модулі CRISPR – Cas (кластеризовані регулярно взаєморозміщені короткі паліндромні повтори - асоційовані з CRISPR білки) є адаптивними системами імунітету, які є в багатьох археях та бактеріях. Ці захисні системи кодуються оперонами, які мають надзвичайно різноманітну архітектуру і високий темп розвитку як для cas гени та унікальний вміст спейсерів. Тут ми пропонуємо оновлений аналіз еволюційних взаємозв’язків між системами CRISPR – Cas та білками Cas. Окреслено три основні типи системи CRISPR – Cas, з подальшим поділом на кілька підтипів та кілька химерних варіантів. З огляду на складність геномних архітектур та надзвичайно динамічну еволюцію систем CRISPR – Cas, уніфікована класифікація цих систем повинна базуватися на безлічі критеріїв. Відповідно, ми пропонуємо „політетичну” класифікацію, яка об’єднує філогенії найпоширеніших cas гени, послідовність та організація повторень CRISPR та архітектура CRISPR–cas локуси.
Параметри доступу
Підпишіться на журнал
Отримайте повний доступ до журналу протягом 1 року
лише 4,60 € за випуск
Усі ціни вказані у нетто-цінах.
ПДВ буде додано пізніше під час оплати.
Оренда або купівля статті
Отримайте обмежений за часом або повний доступ до статей на ReadCube.
Усі ціни вказані у нетто-цінах.
Список літератури
Deveau, H., Garneau, J. E. & Moineau, S. Система CRISPR/Cas та її роль у взаємодії фагів і бактерій. Анну. Преподобний Мікробіол. 64, 475–493 (2010).
Horvath, P. & Barrangou, R. CRISPR/Cas, імунна система бактерій та архей. Наука 327, 167–170 (2010).
Каргінов, Ф. В. і Хеннон, Г. Дж. Система CRISPR: малий захист РНК, керований бактеріями та археями. Мол. Клітинка 37, 7–19 (2010).
Кунін, Є. В. і Макарова, К. С. CRISPR-Cas: система адаптивного імунітету у прокаріотів. F1000 Біол. Респ. 1, 95 (2009).
Sorek, R., Kunin, V. & Hugenholtz, P. CRISPR - широко поширена система, що забезпечує набуту стійкість проти фагів у бактерій та архей. Nature Rev. Microbiol. 6, 181–186 (2008).
van der Oost, J., Jore, M. M., Westra, E. R., Lundgren, M. & Brouns, S. J. Адаптивний та спадковий імунітет на основі CRISPR у прокаріотів. Тенденції Biochem. Наук. 34, 401–407 (2009).
Мойка, Ф. Дж., Діез-Вілласенор, К., Сорія, Е. та Юез, Г. Біологічне значення сімейства регулярно розташованих повторень у геномах архей, бактерій та мітохондрій. Мол. Мікробіол. 36, 244–246 (2000).
Jansen, R., Embden, J. D., Gaastra, W. & Schouls, L. M. Ідентифікація генів, які пов'язані з повторами ДНК у прокаріотів. Мол. Мікробіол. 43, 1565–1575 (2002).
Макарова, К. С., Аравінд, Л., Гришин, Н. В., Рогозін, І. Б. та Коонін, Е. В. Система відновлення ДНК, специфічна для теплолюбних архей та бактерій, передбачена за допомогою аналізу геномного контексту. Нуклеїнові кислоти Res. 30, 482–496 (2002).
Болотін, А., Квінкіс, Б., Сорокін, А. та Ерліх, С. Д. Кластеризовані регулярно переміщені короткі повтори паліндрому (CRISPR) мають розпірки позахромосомного походження. Мікробіологія 151, 2551–2561 (2005).
Мойка, Ф. Дж., Діез-Вілласенор, К., Гарсія-Мартінес, Дж. І Сорія, Е. Втручаючі послідовності регулярно розташованих прокаріотичних повторів походять від чужорідних генетичних елементів. J. Mol. Евол. 60, 174–182 (2005).
Pourcel, C., Salvignol, G. & Vergnaud, G. CRISPR елементи в Yersinia pestis придбати нові повтори шляхом переважного поглинання ДНК бактеріофагів та забезпечити додаткові інструменти для еволюційних досліджень. Мікробіологія 151, 653–663 (2005).
Haft, D. H., Selengut, J., Mongodin, E. F. & Nelson, K. E. У прокаріотичних геномах існує гільдія з 45 сімейств білків, асоційованих з CRISPR (Cas), і безліч підтипів CRISPR/Cas. PLoS Comput. Біол. 1, e60 (2005).
Макарова, К.С., Гришин, Н. Біол. Прямий 1, 7 (2006).
Carthew, R. W. & Sontheimer, E. J. Походження та механізми мікроРНК та siРНК. Клітинка 136, 642–655 (2009).
Barrangou, R. et al. CRISPR забезпечує набуту стійкість до вірусів у прокаріотів. Наука 315, 1709–1712 (2007).
Гаррет, Р. А. та співавт. Імунна система сульфолобалів на основі CRISPR: складність та різноманітність. Біохім. Соц. Транс. 39, 51–57 (2011).
Manica, A., Zebec, Z., Teichmann, D. & Schleper, C. В природних умовах активність опосередкованого CRISPR захисту вірусу в гіпертермофільному археоні. Мол. Мікробіол. 80, 481–491 (2011).
Al-Attar, S., Westra, E. R., van der Oost, J. & Brouns, S. J. Кластеризовані регулярно переміщуються між собою короткі паліндромні повтори (CRISPR): ознака геніального антивірусного захисного механізму у прокаріотів. Біол. Хім. 392, 277–289 (2011).
Гарно, Дж. Е. та співавт. Бактеріальна імунна система CRISPR/Cas розщеплює ДНК бактеріофагів та плазмід. Природа 468, 67–71 (2010).
Sontheimer, E. J. & Marraffini, L. A. Slicer для ДНК. Природа 468, 45–46 (2010).
Мойка, Ф. Дж., Діез-Вілласенор, К., Гарсія-Мартінес, Дж. Мікробіологія 155, 733–740 (2009).
Deveau, H. et al. Фагова реакція на кодований опір CRISPR в Streptococcus thermophilus. Дж. Бактеріол. 190, 1390–1400 (2008).
Brouns, S. J. et al. Невеликі CRISPR-РНК направляють противірусний захист прокаріотів. Наука 321, 960–964 (2008).
Дельчева, Е. та ін. Дозрівання РНК CRISPR шляхом переклад-кодована мала РНК і фактор-хазяїн РНКаза III. Природа 471, 602–607 (2011).
Haurwitz, R. E., Jinek, M., Wiedenheft, B., Zhou, K. & Doudna, J. A. Послідовно-структурна обробка РНК за допомогою ендонуклеази CRISPR. Наука 329, 1355–1358 (2010).
Carte, J., Wang, R., Li, H., Terns, R. M. & Terns, M. P. Cas6 - ендорибонуклеаза, яка генерує провідні РНК для захисту загарбників у прокаріотів. Genes Dev. 22, 3489–3496 (2008).
Хейл, К.Р. та співавт. РНК-кероване розщеплення РНК білковим комплексом CRISPR РНК-Cas. Клітинка 139, 945–956 (2009).
Wang, R., Preamplume, G., Terns, M. P., Terns, R. M. & Li, H. Взаємодія рибоендонуклеази Cas6 з РНК CRISPR: розпізнавання та розщеплення. Структура 19, 257–264 (2011).
Марраффіні, Л. А. та Сонтхаймер, Е. Дж. Самостійність проти несамостійної дискримінації під час спрямованого на РНК-імунітет імунітету CRISPR. Природа 463, 568–571 (2010).
Марраффіні, Л. А. та Сонтхаймер, Е. Дж. Втручання CRISPR обмежує горизонтальний перенос генів у стафілококів шляхом націлювання на ДНК. Наука 322, 1843–1845 (2008).
Wiedenheft, B. et al. Структурні основи активності DNase консервативного білка, причетного до CRISPR-опосередкованого захисту геному. Структура 17, 904–912 (2009).
Білоглазова, Н. та ін. Нове сімейство специфічних для послідовності ендорибонуклеаз, асоційованих з кластеризованими регулярно переміщеними короткими паліндромними повторами. Дж. Біол. Хім. 283, 20361–20371 (2008).
Татусов, Р. Л. та ін. База даних COG: оновлена версія включає еукаріотів. BMC Біоінформатика 4, 41 (2003).
Сінкунас, Т. та ін. Cas3 - це одноцепочечна нуклеаза ДНК та АТФ-залежна геліказа в імунній системі CRISPR/Cas. EMBO J. 30, 1335–1342 (2011).
Soding, J., Remmert, M., Biegert, A. & Lupas, A. N. HHsenser: вичерпний перехідний пошук профілю за допомогою порівняння HMM – HMM. Нуклеїнові кислоти Res. 34, W374 – W378 (2006).
Kleanthous, C. та співавт. Структурно-механістична основа імунітету до ендонуклеазних коліцинів. Структура природи. Біол. 6, 243–252 (1999).
Якубаускас, А., Гедрієне, Дж., Буйніцкі, Дж. М. та Янулайтис, А. Ідентифікація єдиного активного центру HNH в ендонуклеазі рестрикції типу IIS Eco31I. J. Mol. Біол. 370, 157–169 (2007).
Уайт, М. Ф. Структура, функція та еволюція сімейства XPD залізо-сірковмісних 5 ′ → 3 ′ ДНК-геліказ. Біохім. Соц. Транс. 37, 547–551 (2009).
Кунін, В., Сорек, Р. та Гугенгольц, П. Еволюційне збереження послідовностей та вторинних структур у повторах CRISPR. Геном Біол. 8, R61 (2007).
Grissa, I., Vergnaud, G. & Pourcel, C. База даних CRISPRdb та інструменти для відображення CRISPR та створення словників розпірок та повторень. BMC Біоінформатика 8, 172 (2007).
Altschul, S. F. & Koonin, E. V. PSI-BLAST - інструмент для відкриттів у базах даних послідовностей. Тенденції Biochem. Наук. 23, 444–447 (1998).
Марчлер-Бауер, А. і Брайант, С. Х. CD-пошук: анотації білкових доменів на льоту. Нуклеїнові кислоти Res. 32, W327 – W331 (2004).
Макарова, К. С., Вольф, Ю. І. та Коонін, Е. В. Комплексний порівняльно-геномний аналіз токсин-антитоксинних систем типу 2 та пов'язаних з ними рухомих систем реагування на стрес у прокаріотів. Біол. Прямий 4, 19 (2009).
Бабу, М. та ін. Подвійна функція системи CRISPR – Cas у бактеріальному антивірусному імунітеті та відновленні ДНК. Мол. Мікробіол. 79, 484–502 (2011).
Guindon, S. & Gascuel, O. Простий, швидкий і точний алгоритм для оцінки великих філогеній за максимальною ймовірністю. Сист. Біол. 52, 696–704 (2003).
Han, D., Lehmann, K. & Krauss, G. SSO1450 - білок CAS1 від Sulfolobus solfataricus P2 з високою спорідненістю до РНК та ДНК. FEBS Lett. 583, 1928–1932 (2009).
Хан, Д. і Краус, Г. Характеристика ендонуклеази SSO2001 від Sulfolobus solfataricus P2. FEBS Lett. 583, 771–776 (2009).
Гай, К. П., Маєрнік, А. І., Чонг, Дж. П. і Болт, Е. Л. Нова нуклеаза-АТФаза (Nar71) з архей є частиною запропонованої термофільної системи відновлення ДНК. Нуклеїнові кислоти Res. 32, 6176–6186 (2004).
Селенгут, Дж. Д. та ін. TIGRFAMs та властивості генома: інструменти для призначення молекулярної функції та біологічного процесу в геномах прокаріотів. Нуклеїнові кислоти Res. 35, D260 – D264 (2007).
Подяки
Автори дякують М. Тернсу за критичне читання рукопису та корисні дискусії. K.S.M., Y.I.W. та Є.В.К. підтримуються внутрішніми фондами Міністерства охорони здоров'я та соціальних служб США (Національна медична бібліотека); D.H.H. підтримується грантом Національного інституту охорони здоров’я США (1 R01 HG004881); E.C. відзначає фінансування з Університету Умео, Швеція, та Шведської дослідницької ради. С.М. визнає фінансування від Національної ради з питань науки і технічних досліджень Канади (програма Discovery); F.J.M.M. визнає підтримку з боку Університету Аліканте, Іспанія, (Vicerrectorado de Investigacion та Desarrollo e Innovacion) щодо використання його науково-технічних послуг; А.Ф.Й. підтримується урядом Канади через Genome Canada та Інститут геноміки Онтаріо (грант 2009-OGI-ABC-1405). S.J.B. та J.O. підтримуються грантами Veni та TOP від Нідерландської організації з наукових досліджень (NWO).
Інформація про автора
Приналежності
Кіра С. Макарова, Юрій І. Вольф та Євген Васильович Кунін перебувають у Національному центрі біотехнологічної інформації, Національній медичній бібліотеці, Національному інституті охорони здоров’я, 8600 Роквілл Пайк, Бетесда, штат Меріленд 20894, США.
Кіра С. Макарова, Юрій І. Вовк та Євген Васильович Кунін
Даніель Х. Хафт знаходиться в Інституті Дж. Крейга Вентера, 9704 Medical Center Drive, Роквілл, штат Меріленд, 20850, США.
Родольф Баррангу працює в Danisco USA Inc., 3329 Agriculture Drive, Медісон, штат Вісконсин, 53716, США.
Стен Дж. Дж. Браунс і Джон ван дер Уст працюють у лабораторії мікробіології Університету Вагенінгена, Дрейенплейн 10, 6703 HB Вагенінген, Нідерланди.
Стен Дж. Дж. Браунс і Джон ван дер Уст
Кафедра молекулярної біології Еммануель Шарпентьє працює в лабораторії медицини молекулярної інфекції Швеції, Центр мікробних досліджень Умео, Умеоський університет, S-90187 Умео, Швеція.
Філіпп Горват знаходиться у Danisco France SAS, BP10, 86220 Dangé-Saint-Romain, Франція.
Сільвен Моно знаходиться в Департаменті біохімії, Мікробіології та біоінформатики, Факультеті наук та Ені, Університеті Лаваль, Квебек, Квебек G1V 0A6, Канада.
Франциско Дж. М. Мойка знаходиться у департаменті Фізіології, Genética y Microbiología, Університет Аліканте, 03080-Аліканте, Іспанія.
Франциско Дж. М. Мохіка
Олександр Федорович Якунін працює в кафедрі медичних досліджень Бантинга та Кращого, Університет Торонто, Торонто, Онтаріо M5G 1L6, Канада.
Олександр Федорович Якунін
Ви також можете шукати цього автора в PubMed Google Scholar
Ви також можете шукати цього автора в PubMed Google Scholar
Ви також можете шукати цього автора в PubMed Google Scholar
Ви також можете шукати цього автора в PubMed Google Scholar
Ви також можете шукати цього автора в PubMed Google Scholar
Ви також можете шукати цього автора в PubMed Google Scholar
Ви також можете шукати цього автора в PubMed Google Scholar
Ви також можете шукати цього автора в PubMed Google Scholar
Ви також можете шукати цього автора в PubMed Google Scholar
Ви також можете шукати цього автора в PubMed Google Scholar
Ви також можете шукати цього автора в PubMed Google Scholar
Ви також можете шукати цього автора в PubMed Google Scholar
- Голодування в онкології слово обережність Природа Відгуки Рак
- Жувальне шоколадне пряникове печиво - Бейкер від природи
- Суть менеджера; Зміст, цитати, коментарі та відгуки Блог k2p
- ЕСТРОГЕЛ Побічні ефекти, відгуки пацієнтів
- Пропаганда еволюції продуктів харчування для громадської підтримки ГМО Продовольство насамперед