Модифікація аденозину196 метилтрансферазою Mettl3 в 5’-зовнішньому транскрибованому спейсері 47S-пре-рРНК впливає на дозрівання рРНК
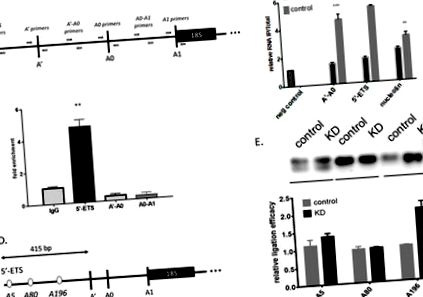
Характеристика взаємодій РНК метилтрансферази Mettl3 з 5′-ETS 47S попередньої рРНК. (A) Схема розташування праймерів RT-qPCR на рРНК до 47S. (B) Відносна кількість фрагментів рРНК в імунопреципітатах, отриманих з антитілами проти m6A з контрольних та клітин Metdl3 KD, кількісно оцінено за допомогою RT-qPCR-аналізу (* ppp ® N6-Methyladenosine Enricment Kit) A'-A0 (праймери для ампліфікації A ' -A0 спейсер), 5′-ETS (праймери для ампліфікації частини 47S попередньої рРНК перед A '), нуклеолін (m6A метильована мРНК на основі даних браузера MeT-DB2.0). (C) Збагачення фрагментів рРНК в імунопреципітататах, отриманих антитілами до Mettl3 з контрольних клітин, кількісно визначеним за допомогою аналізу RT-qPCR. (D) Схема 5′-ETS в рРНК з позначеними аденозинами в контексті передбачуваних послідовностей DRACH. (E) ПЛР-аналіз аналізу лігази Т4 РНК для попередньої рРНК з контрольних та клітин KD Mettl3 (візуалізація фрагментів, розділених за допомогою гель-електрофорезу, та їх відносна кількісна оцінка).
Зміни в обробці попередньої рРНК через 4 дні Mettl3 KD. (A) RT-qPCR аналіз зрілих 18S, 28S та 5.8S рРНК у ядерній та цитоплазматичній фракціях, загальній РНК та зароджуючійся РНК у контрольних та клітинах KD Mettl3. ІРНК Gapdh використовували як контроль цитоплазматичної фракції та загальної РНК, u2 (для ядерної фракції) (* pp B) Аналіз ефективності розщеплення попередньої рРНК за допомогою RT-qPCR в місцях розщеплення A ', A0 (праймери розроблені навколо розщеплення сайт), а також кількісне визначення спейсера РНК A0-A1 у аналізі загальної РНК (* pp C) FISH 5′-ETS 47S попередньої рРНК, спейсер A'-A0 РНК у контролі та клітини KD Mettl3. ДНК фарбували DAPI, а рРНК - міченими Cy3 зондами (таблиця S3). (D) Оцінка швидкості обробки попередньої РНК у місцях розщеплення A0 та A ’у контрольних та клітинах KD Mettl3 за допомогою аналізу актиноміцину D.
Зміни механізмів попередньої обробки РНК після 4 днів застосування Mettl3 KD. (A) Кількісне визначення даних RT-qPCR (* p p B) Вестерн-блот-аналіз факторів обробки рРНК у контрольних та клітинах KD Mettl3. (C) Багаторівневе вирівнювання частини 5′-ETS із модифікованим нуклеотидом m6A (позначеним жовтим) у різних видів. Нуклеотиди послідовності DRACH позначені зеленим кольором.
Прогнозована вторинна структура перших 415 bp 5′-ETS у 47S пре-рРНК. Чорний квадрат позначає спіраль аденозином 196 (наведена чорною стрілкою).
Анотація
Характеристика взаємодій РНК метилтрансферази Mettl3 з 5′-ETS 47S попередньої рРНК. (A) Схема розташування праймерів RT-qPCR на рРНК до 47S. (B) Відносна кількість фрагментів рРНК в імунопреципітатах, отриманих з антитілами проти m6A з контрольних та клітин Metdl3 KD, кількісно оцінено за допомогою RT-qPCR-аналізу (* ppp ® N6-Methyladenosine Enricment Kit) A'-A0 (праймери для ампліфікації A ' -A0 спейсер), 5′-ETS (праймери для ампліфікації частини 47S попередньої рРНК перед A '), нуклеолін (m6A метильована мРНК на основі даних браузера MeT-DB2.0). (C) Збагачення фрагментів рРНК в імунопреципітататах, отриманих антитілами до Mettl3 з контрольних клітин, кількісно визначеним за допомогою аналізу RT-qPCR. (D) Схема 5′-ETS в рРНК з позначеними аденозинами в контексті передбачуваних послідовностей DRACH. (E) ПЛР-аналіз аналізу лігази Т4 РНК для попередньої рРНК контрольних та клітин KD Mettl3 (візуалізація фрагментів, розділених за допомогою гель-електрофорезу, та їх відносна кількісна оцінка).
Зміни в обробці попередньої рРНК через 4 дні Mettl3 KD. (A) RT-qPCR аналіз зрілих 18S, 28S та 5.8S рРНК у ядерній та цитоплазматичній фракціях, загальній РНК та зароджуючійся РНК у контрольних та клітинах KD Mettl3. ІРНК Gapdh використовували як контроль цитоплазматичної фракції та загальної РНК, u2 (для ядерної фракції) (* pp B) Аналіз ефективності розщеплення попередньої рРНК за допомогою RT-qPCR в місцях розщеплення A ', A0 (праймери розроблені навколо розщеплення сайт), а також кількісне визначення спейсера РНК A0-A1 у аналізі загальної РНК (* pp C) FISH 5′-ETS 47S попередньої рРНК, спейсер A'-A0 РНК у контролі та клітини KD Mettl3. ДНК фарбували DAPI, а рРНК - міченими Cy3 зондами (таблиця S3). (D) Оцінка швидкості обробки попередньої РНК у місцях розщеплення A0 та A ’у контрольних та клітинах KD Mettl3 за допомогою аналізу актиноміцину D.
Зміни механізмів попередньої обробки РНК через 4 дні Mettl3 KD. (A) Кількісне визначення даних RT-qPCR (* p p B) Вестерн-блот-аналіз факторів обробки рРНК у контрольних та клітинах KD Mettl3. (C) Вирівнювання декількох послідовностей частини 5′-ETS із модифікованим нуклеотидом m6A (позначеним жовтим) у різних видів. Нуклеотиди послідовності DRACH позначені зеленим кольором.
Прогнозована вторинна структура перших 415 bp 5′-ETS у 47S пре-рРНК. Чорний квадрат позначає спіраль аденозином 196 (наведена чорною стрілкою).
- Безклітинний повнотекстовий дієтичний імуностимулятор CpG модулює мікроРНК біомаркери, пов’язані з
- Безкоштовний повнотекстовий клітинний зв’язок мозок - шкіра та патогенез псоріазу
- Безкоштовне повнотекстове копіювання клітин та введення ядерної оболонки дріжджових мікротрубочок
- Безкоштовний повний текст клітин Нова роль та обіцянки кругових РНК при ожирінні та пов'язаних з ними
- Безкоштовний повнотекстовий клінічний та генетичний аналіз клітин дітей із синдромом Картагенера