Ідентифікація ключових генів та молекулярних механізмів, пов’язаних із низькою несучістю яєць бройлерних племінних курей ad libitum
Анотація
Передумови
Перегодовування знижує продуктивність несучості у курей-розплодів бройлерів, що пов’язано з ожирінням, стеатозом печінки та системним запаленням. Для розкриття основних механізмів, що регулюють вплив режимів годівлі на енергетичний метаболізм та вироблення яєць, був проведений транскриптомічний підхід для скринінгу диференційовано експресованих генів (DEG) у яєчниках, печінці та жировій тканині курчат-бройлерів за умови обмеженого годування.
Результати
Це показало, що в жировій клітці, печінці та яєчниках було виявлено 289, 388 та 204 DEG. Ці DEG були значно збагачені шляхом шляху фагосом, транспорту ліпідів, активності та активності резервуару поживних речовин в яєчниках; біосинтез стероїдних гормонів та метаболізм ксенобіотиків шляхами цитохрому Р450 у жировій тканині; та метаболічні шляхи, активований проліфератором пероксисом рецептор (PPAR) та сигнальний шлях Jak-STAT у печінці. Рецептор естрогену 1, визначений як один з важливих центрів шляхом побудови мережі ІПП, був регульований вгору в групі ad libitum, що дозволило б більшій кількості аполіпопротеїдів переноситися в яєчник.
Висновки
Висока експресія VTG, APOB, CYBB і CTSS в яєчниках призведе до надлишкового відкладення ліпідів, окисного стресу та потенційного пошкодження овуляції. Наші результати сприяють розумінню впливу режимів годівлі на метаболічну регуляцію під час виробництва яєць курчат-розведення бройлерів, а також надають нові докази метаболічної регуляції в результаті інтегрованих багатотканинних процесів.
Передумови
Метою цього дослідження було дослідити гени та їх регуляторну здатність до репродуктивної здатності бройлерів, що залежать від харчування, з цілого рівня транскриптома. У цьому дослідженні досліджували профілі експресії яєчників, печінки та жирової тканини між лібітумом та обмеженим вигодовуванням курей-бройлерів за допомогою технології RNA-seq. Описані тут результати забезпечать значний прогрес у наших знаннях про взаємозв'язок споживання корму, ліпідного обміну та функції яєчників у курей-бройлерів.
Результати
Профілі експресії генів
Ми систематично аналізували дані послідовності з 18 зразків, які включали 3 тканини (жирову, печінкову та яєчникову) від 6 курей (3 кури від групи ad libitum та 3 кури від обмеженої групи годівлі). У середньому 367,3 мільйона необроблених даних було виявлено в 6 зразках з кожної тканини. Після фільтрації приблизно 81% чистих показників у кожному зразку накладали на геном Gallus_gallus-5.0 і використовували для подальшого аналізу експресії генів. У кожному зразку було близько 80% однозначних зчитувань та 1% множинних збігів з геномом (таблиця 1). В середньому було виявлено експресію 12 382, 13 157 та 10 651 гена з жирової тканини, тканин яєчників та печінки відповідно, встановивши поріг кількості зчитування кожного гена вище 1 кількості на мільйон у кожній пробі принаймні однієї групи лікування. Всі ці експресовані гени використовувались для аналізу диференціальної експресії.
Диференціально експресовані гени
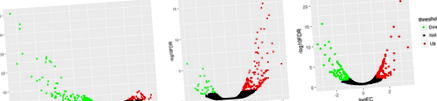
Гени диференціальної експресії аналізували між лібітом та обмеженою групою годування у 3 тканинах (FDR, рис. 1
Онтологія генів та функціональна анотація DEG
На основі анотації DEG, аналіз збагачення кожної тканини з термінів GO біологічного процесу та шляху KEGG були зведені на рис. 2 та Додатковий файл 4: Таблиця S4, встановлення порогу на FDR Рис. 2
GO-аналіз та вихід KEGG-шляхів DEG між лібітом та обмеженою групою годування. (а) Яєчник, (б) жирова тканина, (в) печінка. АТ, біологічний процес; CC, стільниковий компонент; MF, Молекулярна функція
Верифікація експерименту РНК-послідовності
Рівень мРНК DEG, отриманий за допомогою РНК-послідовності з тканин яєчників, печінки та жирової тканини між групами ad libitum та обмеженими групами кормів, додатково вивчали методом qRT-PCR. На малюнку 3 показані рівні експресії десяти генів, випадково вибраних з DEG. Для аналізу RNA-seq вираження CHGB, GAL, SST, APOB, SOST, CHAC1 і SIK1 були регульовані в залежності від групи в півтора-три рази, і експресія ANGPTL4, RRM2 і ACTC1 були регульовані вгору в групі ad libitum у два-чотири рази (рис. 3а). Результати qRT-PCR показали, що експресія цих генів узгоджується з результатами аналізу RNA-Seq, і висока кореляція була виявлена шляхом порівняння результатів двох методів (рис. 3b). Отже, дані qRT-PCR підтвердили, що результати RNA-Seq були надійними.
Порівняльний та кореляційний аналіз диференційовано експресованих генів між qRT-PCR та RNA-seq. (а) Порівняння рівня диференційовано експресованих генів з qRT-PCR та RNA-seq. (b) Кореляція диференційовано експресованих генів між qRT-PCR та RNA-seq. CHGB, хромогранін В; APOB, аполіпопротеїн В; SOST, склеростин; GAL, галанін та GMAP; SST, соматостатин; ACTC1, актин, альфа, серцевий м’яз 1; CHAC1, ChaC-глутатіон-специфічна гамма-глутамілциклотрансфераза 1; SIK1, сольова індукована кіназа 1; ANGPTL4, ангіопоетин типу 4; RRM2, регулятивна субодиниця рибонуклеотидредуктази М2. Експресія гена нормалізувалася як зміна складки за допомогою 18S рибосомна РНК (n = 3 на групу)
Мережа взаємодії білків та білків ДЕГ
Для візуалізації мереж PPI DEG використовували програмне забезпечення Cytoscape (рис. 4). Він складається з 123 вузлів та 159 країв у жировій клітці, 172 вузлів та 373 країв у печінці та 100 вузлів та 153 країв у яєчнику. На малюнку вузол представляє DEG, а край представляє PPI двох DEG. Було 2 (бета-ланцюг фібриногену (FGB), альбумін (ALB)) і 3 гени (ALB, Прото-онкоген Spi-1 (SPI1), плазміноген (PLG)) ідентифіковані як гени-концентратори в жировій клітці та яєчниках із ступенем взаємодії ≥10 відповідно та 24 гени-концентратори у печінці.
Мережа ІЦВ для ДЕГ у жировій тканині, тканинах яєчників та печінки. (а) жировий; (b) Яєчник; (в) Печінка. У мережі різниця ступеня взаємодії відображається кольором. Жовта крапка вказує на те, що вузол має ступінь взаємодії ≥10, який розглядається як гени-концентратори. Чорна крапка вказує на вузол гена диференціальної експресії. ІПП, взаємодія білок/білок; DEG, диференційовано експресовані гени
Обговорення
Обмежене годування бройлерів широко використовується для поліпшення надмірного збільшення маси тіла та зниження ефективності несучості [5,6,7,8]. Згідно з раніше повідомленою інформацією, необмежена годівля курчат-розплодів курчат-бройлерів протягом періоду несучості прискорює дозрівання фолікулів яєчників, що порушує репродуктивну здатність або збільшує невстановлювані яйця [1, 16, 26]. Існує припущення, що причиною необмеженого годування, що погіршує продуктивність яєць курей-розплодників-бройлерів, було головним чином надмірне споживання, що призводило до надмірного споживання енергії [10, 27, 28]. Незважаючи на високі енергетичні потреби, особливо у формуванні жовтка в залежності від метаболізму ліпідів, курей-розплодників-бройлерів мало переносять біологічні речовини до переїдання [19]. Таким чином, варто дослідити основні молекулярні механізми, що регулюють метаболізм ліпідів на продукцію яєць, зокрема щодо впливу харчового статусу [16].
Аналіз STRING у жировій тканині показав, що DEG найбільше збагачують шляхи „біосинтезу стероїдних гормонів” та „метаболізму ксенобіотиків цитохромом P450”, а також молекулярну функцію активності резервуару поживних речовин (додатковий файл 4: Таблиця S4). Жирова тканина є однією з основних ендокринних залоз і вважається одним з основних місць позагонадного синтезу естрогену [43,44,45]. Ми виявили, що ДЕГ у цих двох шляхах переважно регулювались вниз (Додатковий файл 2: Таблиця S2) у групі ad libitum порівняно з групою з обмеженим вигодовуванням. Роль гормональних та метаболічних сигналів жирової тканини потрібно вивчити далі у зв'язку з репродуктивною функцією птиці.
STRING-аналіз у печінці показав, що DEG в основному збагачувались до 9 KEGG-шляхів, оскільки всі ми знаємо метаболічні шляхи, був задіяний сигнальний шлях Jak-STAT та сигнальний шлях PPAR (Додатковий файл 4: Таблиця S4). Крім того, шляхом побудови мережі ІЦВ для ДЕГ з адлібіту проти обмеженого годування було виявлено 24 гени-концентратори (рис. 4). Серед них рецептор естрогену 1 (ESR1 або ER-α) брав участь у метаболічних шляхах. Печінка є головним органом, відповідальним за естрогени, і експресія печінкових естрогенних рецепторів (ER) суворо регулюється репродуктивними функціями та енергетичним гомеостазом [46,47,48]. У печінці синтез білків яєчного жовтка сильно залежить від естрогену та опосередковується ЕР [49]. Синтезовані аполіпопротеїни можуть переноситися через кров до ооцитів, що розвиваються [50]. У нашому дослідженні рівні мРНК ESR1 були регульовані вгору в групі ad libitum порівняно з обмеженою групою годування, що може збільшити синтез попередників білків, таких як VTG II та APOV1, так що прискорене дозрівання фолікулів яєчників та призведе до порушення репродуктивної здатності або збільшення невстановлюваних яєць.
Висновок
Відкладення яєчного жовтка стосуються синтезу, поглинання, транспортування та метаболізму ліпідів в результаті інтегрованих багатотканинних процесів. Ad libitum призведе до надлишку відкладення яєчного жовтка та виробництва дефектних або нерегульованих яєць у курчат-бройлерів. У цьому дослідженні ми визначили ключові ДЕГ, основний біологічний процес та регулюючий шлях шляхом порівняння ad libitum із обмеженим годуванням. Високий рівень ESR1 МРНК у печінці змусить більше аполіпопротеїдів переноситися в яєчник. Висока експресія VTG, APOB, CYBB і CTSS в яєчниках призведе до надлишкового відкладення ліпідів, окисного стресу та потенційного пошкодження овуляції. Усі ці результати можуть виявити молекулярний механізм низької продуктивності яєць бройлерних племінних курей ad libitum. Однак слід дослідити додаткові дослідження щодо ролі гормональних та метаболічних сигналів жирової тканини у зв'язку з репродуктивною функцією у курей-розплодників-бройлерів.
Методи
Управління тваринами та підготовка тканин
Зграя пташенят-плідників для розведення бройлерів Arbor Acres (AA) вирощувалась відповідно до інструкцій керівництва AA Breeder Management, щоб досягти цільової маси тіла до 21 тижня. віку (http://ap.aviagen.com/brands/arbor-acres/). З 22 тиж. у віці сорок пташенят були розділені на дві групи і переведені в окремі клітини для несучок. Одну групу годували дозвільно з раціоном 11,74 МДж/кг енергії, що піддається метаболізму, і 17 г сирого протеїну/100 г корму (група ad libitum), а іншу групу постійно годували за вказаним вище керівництвом (обмежена група). Протягом періоду вирощування доступ до води був необмеженим. Курей годували за графіком освітлення відповідно до рекомендацій компанії-заводчика. Протягом періоду кладки графік освітлення становив 14 год світло, 10 год темно. До 30 тижнів. у віці 3 випадково вибрані з кожної групи курей перед знекропленням знеболювали ізофлураном. Жирові тканини печінки, черевної порожнини та строми яєчників, включаючи дрібні фолікули, були зібрані при розтині. Зразки тканин негайно заморожували у рідкому азоті та зберігали при - 80 ° C для подальшого аналізу транскриптома та qRT-PCR.
Вилучення РНК
Загальну РНК екстрагували з 18 зразків (включаючи жирову клітину, печінку та яєчники, по 6 особин кожної тканини) за допомогою реагенту TRIzol LS (Invitrogen, США). Чистоту та концентрацію РНК оцінювали за допомогою спектрофотометра NanoPhotometer® (IMPLEN, Каліфорнія, США) та набору для аналізу РНК Qubit® у флурометрі Qubi t® 2.0 (Life Technologies, CA, USA). Цілісність РНК перевіряли за допомогою набору для аналізу РНК Nano 6000 системи Bioanalyzer 2100 (Agilent Technologies, Каліфорнія, США), і всі показники цілісності РНК становили більше 9,0.
Підготовка бібліотеки та секвенування транскриптома
Бібліотеки кДНК були побудовані за допомогою NEBNext® Ultra ™ RNA Library Prep Kit для Illumina® (NEB, США). Система Agilent Bioanalyzer 2100 була використана для вимірювання якості бібліотек з 18 зразків. Для послідовності всіх бібліотек була використана платформа Illumina® HiSeq 2500 в Novogene Bioinformatics Technology Co. Ltd. (Пекін, Китай), а довжина зчитування парних кінців становить 125 п.н.
Зчитує контроль якості, вирівнювання та анотацію
Ми використовували FastQC (Версія 0.11.5) для проведення кваліфікованого контролю зчитування необроблених даних [51]. Ми вилучили адаптери та низькоякісні зчитування. Після фільтрування зчитування були зіставлені з еталонним геномом Gallus_gallus-5.0 за допомогою TopHat v2.1.1. [52] включає алгоритм bowtie v2.0.6 [53]. Згодом аналіз проводився в межах R (версія 3.4.0) з використанням пакетів GenomicFeatures [54] для обчислення перекриттів та GenomicAlignments [54] для підрахунку коротких зчитувань, відображених у геномних регіонах на Gallus gallus файл геному GTF.
Ідентифікація гена диференціальної експресії
Диференціально експресовані гени (DEG) аналізували за допомогою пакету R edgeR [55]. Для нормування використовували обрізане середнє значення М-значення (TMM), а для підгонки моделі - узагальнену лінійну модель (від’ємний біном). Гени з коефіцієнтом помилкового виявлення (FDR) Ⓡ Premix Ex Taq ™ II (Tli RNaseH Plus, Takara, Японія) використовували для визначення експресії 10 мРНК. Реакцію qRT-PCR проводили в системі ПЛР у режимі реального часу ABI 7300 (Applied Biosystems, Фостер-Сіті, Каліфорнія). Послідовності праймерів (TSINGKE Biotech, Китай) наведені в додатковому файлі 1: Таблиця S1. Загальний змішаний об’єм 15 мкл ПЛР у кожній лунці містив 7,5 мкл SYBR1 Fast qPCRMix (Takara, Японія), кожен прямий і зворотний праймер 0,8 мкл (10 мкмоль/л), 0,3 мкл еталонного барвника ROX, 1 мкл кДНК-матриці, а решта була Н2О без РНази. Реакцію ПЛР проводили наступним чином: 30 с при 95 ° С, після чого 40 циклів кожен по 5 с при 95 ° С і температура відпалу кожного праймера протягом 34 с і 30 с при 72 ° С. Для розрахунку експресії генів-мішеней використовували метод 2 ΔΔCT, та 18С використовувався як внутрішній контроль.
Аналіз мережі взаємодії білків і білків DEG
Базу даних STRING було проведено для аналізу мережі взаємодії білків/білків (PPI) DEG. Як мінімум один зв’язок з білком та середній показник достовірності (≥0,4) були встановлені як критерії відсічення. Мережі в печінці, жировій клітці або яєчниках були побудовані відповідно до відомої взаємодії курей шляхом вилучення списку генів DEG з бази даних. Для візуалізації мереж PPI використовувалось програмне забезпечення Cytoscape (версія 3.4.0) [57]. Ступінь для кожного гена розраховували шляхом аналізу топологічної структури мережі ІЦВ. Ген зі ступенем ≥10 розглядався як гени-концентратори в мережі.
- Визначення ключових генів та шляхів, пов’язаних із ожирінням у дітей
- Материнські ліпіди під час вагітності пов'язані з підвищеною реактивністю кортизолу у дітей
- Мікробіота товстої кишки людини, пов’язана з дієтою, ожирінням та втратою ваги - PubMed
- Проблеми зі здоров’ям, пов’язані із ожирінням дорослих HealthLink BC
- Гіперфосфатурія, асоційована з гіперкальціурією у хворих на нирковий кальцієвий камінь - Анотація -