LAceModule: Ідентифікація конкуруючих ендогенних РНК-модулів шляхом інтеграції динамічної кореляції
Пов’язані дані
Дані про РНК-послідовності, дані про мікроРНК-послідовності, дані CNV та дані про метилювання генів наведені в Атласі генома раку (TCGA). Вихідні коди LAceModule доступні на https://github.com/GaoLabXDU/LAceModule.
Анотація
Вступ
На відміну від звичайних кореляцій (наприклад, PCC), динамічна кореляція фокусується на зміні кореляції двох змінних після змін у третій змінній (Gunderson and Ho, 2014; Yu, 2018). Наприклад, LA визначається як середнє значення похідної кореляції між двома об'єктами щодо третьої умови (Li, 2002). LA був використаний для ідентифікації генів-кандидатів на захворювання (Li et al., 2007) та генів, пов’язаних із віком людини (Yang et al., 2018), а також для виявлення ключових мікробних видів та факторів середовища мікробної спільноти (Ai et al. ., 2019).
LA є відповідним показником для оцінки кореляційної чутливості цеРНК до мікроРНК. У цьому дослідженні ми спочатку проаналізували ефективність LA у виявленні пар цеРНК. Згодом ми запропонували основу для дослідження модулів ceRNA на основі LA (LAceModule) шляхом інтеграції звичайного PCC та динамічної кореляції LA з множинною невід'ємною факторизацією матриці (NMF). Проводячи подальший аналіз на рак молочної залози, ми виявили, що цеРНК відіграють роль у адгезії клітин, міграції клітин та комунікації між клітинами. Наші результати також показали, що цеРНК можуть представляти перспективні лікарські мішені та маркери для лікування та прогнозу раку.
Результати
LA для прогнозування пар цеРНК
Сучасні дослідження часто використовують PCC або SI для виявлення пар цеРНК. Цей підхід ігнорує чутливість кореляції між РНК до рівнів експресії спільних мікроРНК. Щоб подолати це обмеження, ми використовували LA (Li, 2002) для вимірювання динамічних змін кореляції пари цеРНК залежно від рівнів експресії спільних мікроРНК. Припустимо, що EXPR1 та EXPR2 представляють рівні експресії двох кандидатів на ceRNA R1 та R2, відповідно, тоді як EXPMIC позначає суму рівнів експресії всіх їх загальних мікроРНК, MIC. Ми нормалізували EXPR1 та EXPR2, використовуючи метод оцінювання z, таким чином, що E (EXPR1) = E (EXPR2) = 0, Var (EXPR1) = Var (EXPR2) = 1, де E (·) та Var (·) представляють очікування і дисперсія випадкової величини відповідно.
Припустимо вищесказане, PCC між R1 і R2 є:
LA R1 та R2 щодо їх спільних мікроРНК визначається як LA (R 1, R 2 | MIC) = E (g ′ (EXPMIC)), де g (EXPMIC) = E (EXPR1 × EXPR2 | EXPMIC = expMIC ). Відповідно до леми Штейна (Stein, 1981), якщо сума рівнів експресії всіх спільних мікроРНК MIC відповідає стандартному нормальному розподілу, LA (R1, R2 | MIC) = E (EXPR1 × EXPR2 × EXPMIC), обчислення LA можна спростити, як показано нижче:
де N - кількість вибірки. Ми виконали перетворення даних на EXPMIC, використовуючи метод Ван дер Вардена, щоб переконатися, що EXPMIC відповідає стандартному нормальному розподілу. Для EXPMIC1, EXPMIC2, ⋯, EXPMICN ми спочатку отримали їх ранги r1, r2, ⋯, rN, а згодом обчислили перетворене значення наступним чином:
де Φ (·) - кумулятивна функція розподілу стандартного нормального розподілу.
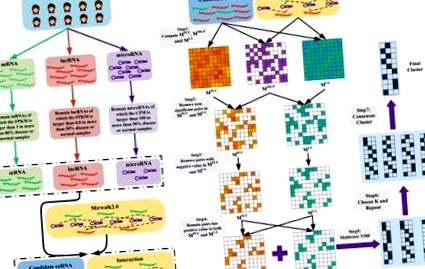
(A) Підготовка даних. Ми отримали дані РНК-послідовності мРНК та lncRNAs, а також дані microRNA-seq мікроРНК. Згодом ми видалили неекспресовану та слабо експресовану РНК. Нарешті, ми зберегли РНК, які були представлені в наборах даних взаємодії РНК-мікроРНК (тут Mirwalk2.0) як кандидати-цеРНК. (B) Огляд LAceModule. Вхідні дані LAceModule - це кандидатні профілі експресії ceRNA, профілі експресії microRNA та взаємодії RNA-microRNA. Для кожної пари цеРНК-кандидатів можна отримати значення PCC, значення LA та ступінь значущості значення спільних мікроРНК (MS-P). Для пар з вищими значеннями MS-P (поріг 0,05) негативні значення PCC або значення LA повинні бути видалені (тобто значення PCC та значення LA цих пар встановлюються на нуль). Мультипрозорий NMF виконується з використанням матриці PCC, матриці LA та різних K як входів. Кращий К вибирається шляхом порівняння чотирьох метрик оцінки кластеризації. Згодом процедури NMF із декількома переглядами повторюються 10 разів із найкращими K та різними початковими значеннями. Остаточні модулі отримані шляхом консенсус-кластеризації результатів повторення.
Таблиця 1
Значення LA, PCC та SI валідованих пар цеРНК.
| ENSG00000234741 | ENSG00000171862 | −0,058 | 0,040 | −0,008 | 0,005 | BRCA |
| ENSG00000251562 | ENSG00000070831 | 0,043 | −0,009 | 0,002 | 0,001 | BRCA |
| ENSG00000251562 | ENSG00000135446 | −0,377 | 0,000 | −0,003 | 0,022 | BRCA |
| ENSG00000115414 | ENSG00000026508 | 0,082 | −0,003 | −0,001 | 0,001 | BRCA |
| ENSG00000108821 | ENSG00000026508 | −0,014 | 0,082 | 0,001 | 0,029 | BRCA |
| ENSG00000171862 | ENSG00000038427 | 0,379 | 0,075 | −0,004 | 0,002 | BRCA |
| ENSG00000038427 | ENSG00000139687 | 0,368 | 0,058 | 0,000 | 0,003 | BRCA |
| ENSG00000226950 | ENSG00000168036 | 0,131 | 0,103 | −0,003 | 0,012 | LIHC |
| ENSG00000234741 | ENSG00000150593 | 0,205 | −0.205 | −0,014 | 0,003 | LIHC |
| ENSG00000234741 | ENSG00000171862 | −0,003 | −0.107 | −0,002 | 0,013 | LIHC |
| ENSG00000241388 | ENSG00000057663 | 0,035 | −0,068 | −0,005 | 0,033 | LIHC |
| ENSG00000251164 | ENSG00000148516 | −0,093 | 0,097 | −0,001 | 0,004 | LIHC |
| ENSG00000251164 | ENSG00000168615 | −0,392 | 0,411 | 0,003 | 0,034 | LIHC |
(A) Значення AUC для прогнозування пари цеРНК з LA, PCC та SI у BRCA та LIHC. (B) Кластерна оцінка трьох методів на різних матрицях у BRCA. (C) Кластерна оцінка трьох методів на різних матрицях в LIHC. (D) Порівняння ентропії зміни згину гена в модулях між різними методами кластеризації. (E) Порівняння середньої валідованої мікроРНК кожної пари в модулях між різними методами. (F) Порівняння дисперсії дисрегульованих генів у модулях між різними методами. Верхній ряд: ігнорування напрямку порушення регулювання, нижній ряд: врахування напрямку порушення регулювання. (* p Малюнок 1B), структура, заснована на багатовиробничому NMF (Liu et al., 2013) для систематичної ідентифікації модулів ceRNA за допомогою LA. Для кожної пари кандидатів ceRNA ми розрахували значення PCC, значення LA та ступінь значущості спільних мікроРНК (MS-P) (див. Розділ Матеріали та методи) для побудови трьох матриць M PCC, M LA та M MS − P відповідно. Згодом, коли значення MS-P кандидатних пар цеРНК становили ≥ 0,05, ми встановлюємо їх відповідні значення PCC та значення LA рівними нулю. Завдяки вимозі щодо негативного негативного впливу в рамках багатофункціонального перегляду NMF, ми встановлюємо нульові значення в M PCC та M LA. Враховуючи, що пара ceRNA повинна бути коекспресована та чутлива до змін у експресії їх спільних мікроРНК, ми встановлюємо значення в тому самому вході M PCC та M LA кандидатних пар ceRNA до нуля, якщо будь-яке з цих значень дорівнює нулю. Нарешті, ми інтегрували M PCC та M LA, використовуючи багатопрофільний NMF для ідентифікації модулів ceRNA.
Для багаторівневого NMF є два види спостереження M = M PCC, M LA>, кожен з яких є G × G невід’ємною матрицею, де G - кількість кандидатів на ceRNA. Кожну матрицю в M, M v ∈ M PCC, M LA> можна розкласти на фактори UG × K v ≥ 0 та (VG × K v) T ≥ 0, що M v ≈ U v (V v) T і кожен рядок (V v) T можна розглядати як представлення K-рангу відповідної точки-кандидата ceRNA. Тут ми спробували визначити представлення низького рангу, яке підходить для обох подань, яке визначається як (V *) T. Ми розклали кожну матрицю на M і розробили кожну (V v) T якомога ближче до (V *) T. Тому ми визначили цільову функцію наступним чином:
де λPCC та λLA налаштовують відносну вагу серед різних поглядів та між стандартною похибкою ЯМФ та розбіжностями між (V *) T, (V PCC) T та (V LA) T. Ми використовували ітераційну процедуру шляхом оновлення однієї змінної, зберігаючи решту змінних, зафіксованих для вирішення цієї задачі оптимізації (див. Деталі в розділі Матеріали та методи). Після обчислення (V *) T, ми отримали мітку модуля РНК i, використовуючи r g max j = 1, 2, ⋯, K V i j * .
Зауважимо, LAceModule вимагає попереднього визначення кількості модулів, K. Ми оцінили ефективність кластеризації, щоб вибрати оптимальний K в діапазоні від 10 до 400 з кроком в 10, розглянувши чотири показники (Рисунки 2B, C), а саме C-індекс (Hubert and Schultz, 1976), McClain-Rao (McClain and Rao, 1975), точковий двобічний коефіцієнт кореляції (Milligan, 1981) та силуетний коефіцієнт (Rousseeuw, 1987). Одночасно розглядаючи чотири метрики на двох матрицях, ми вибрали K = 360 у BRCA та K = 370 у LIHC. Щоб отримати надійні модулі ceRNA, LAceModule повторив процедури перегляду NMF із декількома переглядами і обчислив матрицю консенсусу для ідентифікації модулів ceRNA, використовуючи кластерний алгоритм розподілу подібності (CSPA) (Strehl and Ghosh, 2003). Зокрема, CSPA генерує двійкову матрицю для кожного результату кластеризації NMF із декількома переглядами, при цьому “1” представляє два асоційовані гени в одному кластері, а “0” - для не. Матриця консенсусу - це сума цих двійкових матриць. Модулі ceRNA можна ідентифікувати за допомогою спектральної кластеризації на цій матриці консенсусу, використовуючи оптимальний K, вибраний вище.
Порівняння методів LAceModule та PCC/SI
Ми використовували NMF для заміни багатовидового NMF та матриці PCC або матриці SI як вхідні дані для порівняння продуктивності звичайних та динамічних кореляцій при виявленні модулів ceRNA. У матриці PCC та матриці SI негативні значення або відповідні значення MS-P ≥ 0,05 були встановлені на нуль. Ми також протестували K в діапазоні 10–400 з кроком в 10 і оцінили ефективність кластеризації за тими ж показниками, що зазначені в Розділі Ідентифікація модулів ceRNA, використовуючи LAceModule. Ми вибрали Ks, що дорівнюють 350 та 360 для результатів на основі PCC та SI на основі BRCA, тоді як K, що дорівнюють 360 та 340, відповідно, були обрані для LIHC (Рисунки 2B, C). У наступних розділах ми використовували «PCC + LA» для представлення модулів, виявлених LAceModule, а також «PCC» та «SI» для представлення модулів на основі PCC або SI, відповідно.
ЦеРНК регулюються за допомогою спільних мікроРНК. Отже, модулі ceRNA можуть мати тенденцію ділити більше мікроРНК у кожній парі. Ми використовували експериментально підтверджену взаємодію мРНК-мікроРНК у miRTarBase (Chou et al., 2016), щоб оцінити середню кількість спільних мікроРНК у парі. Результати показані на малюнку 2Е. Модулі “PCC + LA” мали в середньому більше мікроРНК в порівнянні з модулями “PCC” (FDR = 1,84E-02 у BRCA, FDR = 1,84E-02 у LIHC; однобічний тест Вілкоксона) та “SI” (FDR) = 1,05E-06 у BRCA, FDR = 2,62E-09 у LIHC; однобічний тест Вілкоксона). Більше того, модулі «PCC» мали в середньому більше мікроРНК, ніж модулі «SI» (FDR = 8,46E-03 у BRCA, FDR = 3,82E-05 у LIHC; однобічний тест Вілкоксона).
У сукупності порівняння змін складки генів, коефіцієнт дисрегуляції генів та кількість спільних мікроРНК свідчать про те, що інтеграція звичайних та динамічних кореляцій забезпечує краще виявлення модулів ceRNA, ніж лише звичайна кореляція.
Функціональний аналіз модулів ceRNA при раку молочної залози
- Ідентифікація та кількісна оцінка компонентів аромату характеру у свіжому козячому сирі у стилі Шевре
- Ідентифікація відбитка мозку на надмірну вагу та ожиріння - ScienceDirect
- Профілі ідентифікації та підпису для медіаторів ліпідів, що сприяють розсмоктуванню, у людини
- Виявлення ожиріння у пацієнтів з діабетом 2 типу від діабету первинної медичної допомоги в Австралії
- Факти кліща; Ідентифікація, контроль; Профілактика