Ідентифікація потенційних біомаркерів та шляхів розвитку виразкового коліту з комбінованим аналізом даних мікрочипів експресії загальної мРНК та мікроРНК
Lili Yang 1,2 #, Yaoyao Bian 3 #, Zhengjun Li 4 #, Yan Yan 5, Junyi Li 1, Wenlin Li 2, Li Zeng 1,2
Внески: (I) Концепція та дизайн: L Yang, Y Bian; (II) Адміністративна підтримка: відсутні; (III) Надання навчальних матеріалів або пацієнтів: відсутні; (IV) Збір та збір даних: Y Bian; (V) Аналіз та інтерпретація даних: Z Li, J Li; (VI) Написання рукописів: Усі автори; (VII) Остаточне затвердження рукопису: Усі автори.
# Ці автори зробили однаковий внесок у цю роботу.
Передумови: Виразковий коліт (UC) - це хронічне, рецидивуюче та неспецифічне запальне захворювання, яке включає різні гени та шляхи їх патогенезу. Все більше доказів показали, що мікроРНК (miРНК) діють як ключові посттранскрипційні регулятори експресії генів в UC. Це поточне дослідження мало на меті виявити ключові мікроРНК, потенційні цільові гени та відповідні шляхи, задіяні в UC, щоб розкрити їх основні молекулярні механізми за допомогою біоінформатичного аналізу.
Методи: Профілі експресії мРНК та міРНК були отримані та завантажені з бази даних Gene Expression Omnibus (GEO). Диференціально експресовані гени (DEG) та мікроРНК (DEMI) були отримані за допомогою програмного пакету R.
Результати: Всього було отримано 79 DEG та 47 DEMI. І була виявлена група міРНК та їх цільові мРНК. Це показало, що miR-1231 може бути ключовим регулятором для DUOX2 і TFF1. CCL11 може бути потенційно націлений на miR-625. MMP1 може грати життєво важливу роль у розвитку UC, регулюючи шлях передачі сигналів miR-1228/PPAR. Крім того, ми попередньо перевірили найбільш виражені вгору/вниз мікроРНК (miR-92b, miR-625) та дві відповідні їм цільові мРНК (AQP8 та TAGAP, CCL11 та CHI3L1) у тканинах товстої кишки моделей UC. Результати відповідали аналізу мікрочипів.
Висновки: Ці висновки можуть дати нові уявлення про представлення ключових механізмів, пов'язаних з розвитком UC.
Ключові слова: Виразковий коліт (UC); мікроРНК (miРНК); диференційовано експресовані гени (DEG); аналіз біоінформатики; аналіз функціонального збагачення
Подано 06 березня 2019 р. Прийнято до публікації 03 червня 2019 р.
Вступ
Виразковий коліт (UC), підвид запального захворювання кишечника (IBD), є хронічним, рецидивуючим і неспецифічним запальним захворюванням, яке обмежується слизовою і підслизовою оболонками прямої кишки або товстої кишки. Характеризується двома періодами активного захворювання та ремісії. Типовими клінічними проявами є діарея, гнійний стілець та біль у животі (1).
UC вражає мільйони людей у всьому світі. Найвища захворюваність на UC у Північній Америці та Північній Європі становила 6–15,6 та 10–20,3 випадків відповідно на 100 000 щорічно (2). Недавній оглядний аналіз 44 досліджень, які включали 31 287 азіатських пацієнтів, виявив 0,85% показника поширеності UC (3). Крім того, пацієнти, які страждають на UC, мають високий ризик розвитку колоректального раку (CRC) (4). UC приніс значний особистий та суспільний тягар. Крім того, активний UC може знизити фізичну та психічну якість життя та посилити психологічний дистрес.
У цьому дослідженні ми отримали набори даних про мікрочипи експресії мРНК та мікроРНК з Онібусу генної експресії (GEO) та визначили групу ключових мікроРНК та потенційно цільові гени, задіяні в UC, за допомогою біоінформатичного аналізу. Крім того, були відібрані та найефективніші мікроРНК та їх цільові гени та проведені для попередньої перевірки за допомогою qPCR у реальному часі (qRT-PCR). Це дослідження мало на меті запропонувати підписи miRNA, корисні для активного виявлення та діагностики UC, а також дослідити основний патогенез шляхом виявлення потенційних mRNA-орієнтованих mRNA на молекулярному рівні.
Методи
Критерії збору та включення досліджень
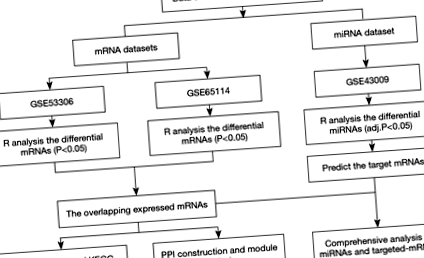
Набори даних профілю виразу мікрочипів мРНК були отримані та завантажені з бази даних GEO (доступна в Інтернеті: http://www.ncbi.nlm.nih.gov/geo) шляхом пошуку за такими ключовими словами: 'РНК', 'активний виразковий коліт', та 'Homo sapiens' (організм). Критеріями включення були такі: (I) тканини товстої кишки у дорослих пацієнтів з активним UC (не клітинами); (II) зразки в групі UC без отримання будь-яких втручань та лікування; та (III) як кількість UC, так і зразок контролю стану здоров'я ≥12. Крім того, у наборах даних профілю miRNA здійснювали пошук за допомогою ключових слів: 'miRNA', 'активний виразковий коліт' та 'Homo sapiens' (організм) згідно з наступними критеріями: (I) тканини товстої кишки у активних хворих на UC (не клітини) та (II) зразки в групі UC без будь-яких втручань та лікування. Після скринінгу було відібрано два набори даних про експресію мРНК [GSE53306 (16) та GSE65114], та отримано один аналіз набору експресії мікроРНК (GSE43009). Процес обробки та аналізу даних представлений на рисунку 1.
Дані мікрочипів
У цьому дослідженні платформа для GSE53306 була заснована на експресійному біччіпі GPL14951 Illumina HumanHT-12 WG-DASL V4.0 R2, який складався з 16 активних зразків UC та 12 контролів. Платформою для GSE65114 був GPL16686 (HuGene-2_0-st) Affymetrix людський ген 2.0 ST масив, який включав 16 активних зразків UC та 12 контролів. Платформа для GSE43009 була заснована на багатовидовому масиві miRNA-3 Affymetrix GPL16384, який складався з п'яти контролів та п'яти зразків UC.
Обробка даних та ідентифікація диференційовано експресованих генів (DEG)/диференціально експресованих мікроРНК (DEMI)
Вихідні дані були завантажені з бази даних GEO, а потім нормалізовані та стандартизовані за допомогою програмного пакету R. Аналіз диференціальної експресії генів проводили через пакети лімми в упаковці Bioconductor (17) (доступно в Інтернеті: http://www.bioconductor.org/). Теплові карти двох наборів даних мРНК наносили на карту за допомогою пакета gplots у R, щоб візуалізувати значення експресії генів у різних зразках. Коли ми вибрали DEG, P 1 розглядали як граничні значення, де FC - кратна зміна. Значні DEMI були перевірені шляхом зустрічі обох присвоєних. P 1. Інтернет-інструмент Venny (доступний в Інтернеті: http://bioinfogp.cnb.csic.es/tools/venny) був використаний для ідентифікації DEG у двох наборах даних мРНК. Визначені DEMI були збережені для подальшого біоінформатичного аналізу.
Аналіз функціонального збагачення DEG
Аналіз збагачення генної онтології (GO), який використовується для анотування генів, проводився для визначення суттєво регульованих функцій, тобто біологічних процесів (BP), клітинного компонента (CC) та молекулярної функції (MF). Для представлення систематичного аналізу, анотації та візуалізації функцій генів був проведений аналіз збагачення Кіотської енциклопедії генів і геномів (KEGG). Збагачення GO та аналіз шляхів KEGG проводились з використанням бази даних для анотацій, візуалізації та інтегрованого відкриття (DAVID) (доступна в Інтернеті: http://david.abcc.ncifcrf.gov/) для виявлення біологічної значущості генів при P 0,4 визначається як критерій відсічення. Потім інтегровані регуляторні мережі візуалізував Cytoscape (18). Нарешті, плагін Molecular Complex Detection (MCODE) був застосований для екранування модулів мережі PPI.
Прогнозування мішеней міРНК
Відповідні цілі miRNA прогнозували за допомогою miRWalk (доступно в Інтернеті: http://zmf.umm.uni-heidelberg.de/apps/zmf/mirwalk2/), що є всеосяжним атласом прогнозованих та підтверджених взаємодій міРНК-мішень. Потенційні мішені мікроРНК були визначені принаймні за чотирма програмами.
Тварини та експериментальний дизайн
Двадцять самців щурів SD (180 ± 20 г) у віці 8–10 тижнів були придбані у Дослідному племінному господарстві Цинлуншань (Нанкін, Китай). Тварин утримували у стандартних умовах із температурою навколишнього середовища 22 ± 2 ° C при 12 год світло/темрява. Щурів з необмеженим доступом до стандартного чау-чау випадковим чином розподіляли на дві групи (по десять щурів на групу). Щурів гострого коліту викликали даючи питну воду, що містить 3,5% (мас./Об.) Декстрану сульфату натрію (DSS) (Sigma-Aldrich, США), тоді як контролі отримували питну воду без DSS. Через 10 днів усіх щурів забивали під наркозом із ізофлураном, а тканини товстої кишки збирали для подальшого аналізу. Всі проведені протоколи на тваринах відповідали китайським Правилам розміщення та догляду за тваринами, сформульованим та схваленим Нанкінським університетом китайської медицини.
Екстракція РНК та qRT-ПЛР
Відповідно до вказівок виробника, загальну РНК тканин товстої кишки в обох групах UC та контролях витягували за допомогою реагенту TRIzol (Invitrogen, Каліфорнія, США). Праймери були отримані з Genscript, а послідовності представлені в таблиці S1. Експресія мРНК та міРНК нормалізувалася щодо експресії GAPDH та U6, відповідно.
Фарбування гематоксиліном та еозином (ВІН)
Тканини товстої кишки розрізали на ділянку товщиною 4 мкм після фіксації, зневоднення та вкладання, а потім фарбували гематоксиліном та еозином. Три випадкові зрізи кожної тканини були зображені під мікроскопом (Leica, німецька).
Статистичний аналіз
Статистичний аналіз проводили за допомогою програмного забезпечення SPSS 19.0. Усі дані виражали як середнє значення ± стандартне відхилення (SD) та аналізували за допомогою Graph Pad Prism (Версія 5.0, Inc., Каліфорнія, США). Статистичне порівняння в двох групах проводили неспареними t-тестами Стьюдента. А значення Р менше 0,05 вважалося значущим.
Результати
Ідентифікація DEG
Два набори даних мікрочипів GSE53306 та GSE65114 були нормалізовані, як показано на малюнку 2. Всього з набору даних GSE53306 було продемонстровано 1052 DEG. Крім того, з набору даних GSE65114 було виявлено 296 DEG. Ієрархічні кластерні теплові карти DEG в обох наборах даних представлені на рисунку 3. А 79 генів диференційовано виражені в обох двох наборах даних.
Аналіз функціонального та збагачувального шляхів DEG
79 накладених вище генів були завантажені в інструмент онлайн-аналізу DAVID для функціонального призначення та збагачення шляху зі значенням Р 0,4. Ми виявили кілька генів-концентраторів, такі як інгібітор металопептидази 1 (TIMP1), мотив CXC-хемокіновий ліганд 10 (CXCL10), мотив CXC-хемокіновий ліганд 13 (CXCL13), CC-мотив-хемокіновий ліганд 11 (CCL11), CC-мотив-хемокіновий ліганд 19 (CCL19) та виберіть селектин L (ПРОДАЙТЕ).
Згодом 55 отриманих пар ІПЦ пройшли аналіз за допомогою Cytoscape для відображення складних взаємозв’язків. Більше того, було виявлено два кластери з модуля DCOME, який складався з дев'яти генів (малюнок 5B), SELL, CCL19, CXCL13, CXCL10, CCL11, регуляторної субодиниці рибонуклеотидредуктази M2 (RRM2), PDZ-зв'язуючої кінази (PBK), центросомного білка 55 (CEP55) та взаємодія рецепторів гормонів щитовидної залози 13 (TRIP13). Нарешті, ми виявили, що ці гени були збагачені імунною відповіддю DAVID.
Прогнозування DEMI та виявлення потенційних генів-мішеней
Для ідентифікації DEMI використовувався набір даних профілю GSE43009. Після попередньої обробки даних ми усунули одну групу в вибірці UC та контрольній вибірці через поганий ієрархічний кластер. Всього було виявлено 47 DEMI. Далі, використовуючи базу даних miRWalk, були отримані прогнозовані цілі miRNA. Перекриваються мРНК, передбачені miRWalk та DEG, визначені вище, представлені в таблиці 3. Ми виявили, що miR-92b та miR-625 були найбільш значущими miРНК. PTGIS передбачали як потенційну мішень чотирьох miРНК, а саме miR-1228, miR-1268, miR-1231 та miR-92b. DES був визначений як потенційна мішень трьох miRNAs, а саме, miR-939, miR-1268 та miR-1226. UNC13D було визнано потенційною мішенню miR-939, miR-1268 та miR-1908. Більше того, сім генів (у тому числі MMP10, DPP10 та PCK1) потенційно мішенню міР-92b, тоді як сім мРНК (включаючи CCL11) потенційно мішенню міР-625.
Перевірка міРНК та цільових мРНК за допомогою qRT-PCR
Щоб продемонструвати миРНК та цільові мРНК, які ми ідентифікували, ми вибрали найбільш суттєво виражену вгору/вниз міРНК (miR-92b та miR-625) та вибрали дві відповідні їм цільові мРНК (AQP8 та TAGAP, CCL11 та CHI3L1) на основі результатів аналізу біоінформатики та попередніх літератур для перевірки в тканинах товстої кишки на моделях UC гризунів. Фарбування ВІН індукованого DSS UC та контроль (рис. 6) запропонували успішну підготовку моделі. Результати qRT-ПЛР показали, що міРНК та відповідні мРНК були суттєво виражені (P
Висновки
У цьому дослідженні із наборів даних GEO було виявлено загалом 79 DEG та 47 DEMI, що може надати початкові докази того, що ці гени та мікроРНК можуть служити потенційними сигнатурами, пов'язаними з активним UC. Генезис UC - надзвичайно складний процес, під час якого відбувається багато генетичних та епігенетичних модифікацій рушійних генів.
Серед визначених нами генів-концентраторів CXCL10 був виділений як найвищий ступінь генів зв’язку. CXCL10, а також CXCL13 повідомляли про значне збільшення хворих на ВЗК (19). TIMP1 є членом сімейства генів TIMP і кодує матриксні металопротеїнази (ММР), які є ключовими ефекторами опосередкованих тканинами травм Т-клітин. Обидва гени беруть участь у запальній реакції після пошкодження тканин та відновлення при ВЗК. TIMP1 виявлено у щурів із хворобою Крона (20). Інше дослідження (21) на основі нокаутованих мишей показало, що дефіцит TIMP1 може спричинити високу експресію імуногенних генів. CCL19, як член цитокінів CC, суттєво виражається у хворобі Крона (CD) і може нормально призводити до мікросередовища хемокінів (22). У світлі вищенаведених результатів ми також припустили, що ці гени можуть представляти біомаркери-кандидати для UC.
Однак це дослідження має деякі обмеження. По-перше, результати були отримані із загальнодоступної бази даних мікрочипів GEO, а платформи аналізу трьох наборів даних GSE були неоднаковими. По-друге, зразків було обмежено, що може спричинити достовірність нашого висновку. Для підтвердження наших результатів необхідні подальші дослідження з більшою кількістю зразків та уніфікованою технологічною платформою виявлення.
У сукупності в нашому поточному дослідженні використовувався комплексний біоінформатичний аналіз для визначення експресії мРНК та мікроРНК між активним UC та контролем. Була ідентифікована група мікроРНК та їх цільові гени, і кілька з них були попередньо підтверджені на моделі гризунів, яка може служити потенційними біомаркерами, пов'язаними з UC. Крім того, ми виявили кілька важливих генних функцій та шляхів, які можуть допомогти нам зрозуміти молекулярні механізми UC. Однак необхідні подальші експериментальні та функціональні дослідження, щоб визначити точну роль та механізми UC.
Подяка
Фінансування: Це дослідження було підтримане грантами Національного фонду природничих наук Китаю (грант № 81704084, 81673982 та 81603529), науково-технічних проектів Бюро традиційної китайської медицини провінції Цзянсу (YB2017002 та YB2015002), Природничих наук Заснування вищих навчальних закладів провінції Цзянсу (16KJB360002), Програми післядипломної дослідницької та практичної діяльності провінції Цзянсу (KYCX18_1541), що фінансується проектом Qing Lan, проектом фінансування дисципліни в галузі китайської медицини та Китайською стипендіальною радою (CSC).
Виноска
Конфлікт інтересів: Автори не мають заявляти про конфлікт інтересів.
Етична заява: Автори несуть відповідальність за всі аспекти роботи, забезпечуючи належне дослідження та вирішення питань, що стосуються точності або цілісності будь-якої частини роботи.
- ВЗК Крона; s Хвороба Виразковий коліт; Дієта ВППБ, частина 5 з 5; Цільнопродовольча рослинна дієта
- Як я контролював свій виразковий коліт
- Ідентифікація слинних та плазмових біомаркерів на ожиріння у дітей за нецільовим методом
- Чи дієтичне волокно добре для пацієнтів з Кроном; s Хвороба або виразковий коліт Блог журналів AGA
- Визначення ключових генів та шляхів, пов’язаних із ожирінням у дітей