Одноклітинна РНК-послідовність виявляє зміни, спричинені ожирінням, у мікросередовищі молочної залози з мутацією Brca1
Пов’язані дані
Анотація
1. Вступ
Ожиріння було визнано провідною проблемою охорони здоров'я у всьому світі [1]. Приблизно дві третини американців мають надлишкову вагу (Індекс маси тіла (ІМТ) ≥ 25 кг/м2), і понад 50% цих людей із надмірною вагою страждають ожирінням (ІМТ ≥ 30 кг/м2) [2]. Збільшення кількості велико когортних досліджень продемонструвало, що ожиріння схильне до ризику захворювань, зокрема раку [1,2,3]. Широкомасштабні епідеміологічні дослідження також показали, що жінки з ожирінням мають більший рівень захворюваності на рак молочної залози, а пацієнти з ожирінням молочної залози, як правило, мають поганий прогноз [4,5,6,7,8]. Було виявлено безліч механізмів, що пояснюють кореляцію між ожирінням та раком молочної залози, такі як індуковані ожирінням зміни жирової ендокринної функції, системний імунітет та метаболічний гомеостаз [9,10,11,12,13]. Тим не менше, точні механізми, що лежать в основі опосередкованого ожирінням прогресування раку молочної залози, залишаються неповними.
2. Результати
2.1. Дієти з високим вмістом жиру викликають ожиріння у Brca1 -/-; p53 +/− Миші для аналізу scRNA-seq
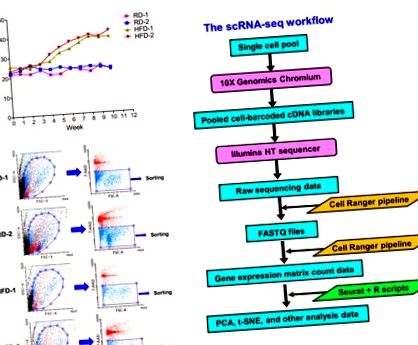
Індукувати Brca1 -/-; p53 +/− мишей для розвитку ожиріння, ми годували двомісячних самок мишей дієтами з високим вмістом жиру (HFD) протягом 10 тижнів. Самки мишей подібного віку в групі з нормальною вагою годувались регулярними дієтами (РД) протягом того самого періоду часу. Під час дієтичного годування ми щотижня вимірювали їх вагу. Рисунок 1 А показує, що миші, які харчувалися дієтами з високим вмістом жиру, значно набирали вагу до середніх 40,11 ± 2,14 г через 10 тижнів порівняно з контрольною групою мишей із середньою вагою 22,33 ± 0,52 г. В кінці експериментів з дієтичним вигодовуванням експериментальних мишей евтаназували, а молочні залози збирали для одержання одноклітинних препаратів, як описано в "Матеріали та методи". У цьому дослідженні ми зосередилися лише на впливі ожиріння, спричиненого HFD, на стромальні фібробласти та імунні клітини. Для збагачення цих типів клітин ми використовували анти-EpCAM антитіло для видалення епітеліальних клітин молочної залози. Збагачені неепітеліальні клітинні фракції фарбували барвником 7-AAD, а потім піддавали сортуванню клітин для виділення життєздатних поодиноких клітин (рис. 1 Б). Враховуючи, що мертві клітини забарвлювали 7-AAD, 7-AAD-негативні клітини відводили для сортування клітин для очищення життєздатних поодиноких клітин (Малюнок 1 B). Відсортовані життєздатні поодинокі клітини піддавали аналізу scRNA-seq з використанням системи 10 × Genomics Chromium.
Аналіз scRNA-seq, відсортованих клітинами з сортуванням клітин (FACS), виділених із молочних залоз HFD або RD Brca1 -/-; p53 +/− миші. (A) Годування дієтами з високим вмістом жиру, спричиненими ожирінням у Brca1 -/-; p53 +/− миші. Дані про масу тіла експериментальних мишей були побудовані на основі інформації за тиждень. Чотири самки C57BL6 Brca1 -/-; В експерименті брали участь миші p53 +/− (група звичайної дієти: RD-1 та RD-2; група з високим вмістом жиру: HFD-1 та HFD-2). (B) FACS сортування життєздатних клітин молочної залози. Збагачені неепітеліальні клітини фарбували барвником 7-AAD, а потім сортували, використовуючи стан закритого типу, показаний у даних FACS. 7-AAD-негативні клітини були закриті для сортування життєздатних клітин. (C.) Діаграма робочого процесу scRNA-seq.
Як показано на малюнку 1 С, технологічним дизайном системи 10 × Genomics Chromium для scRNA-seq є мікрофлюїди на основі крапель з мікроканалами для генерування крапель водної олії-емульсії [30]. Кожна краплина ідеально складається з реагенту для зворотної транскрипції (RT), однієї клітини та кульки, кон'югованої з олігонуклеотидами. Послідовність олігонуклеотидів має універсальну послідовність адаптерів, призначену для ампліфікації ПЛР, послідовність клітинних штрих-кодів, специфічну для кожної гранулки, послідовність унікального молекулярного ідентифікатора (UMI) для послідовного аналізу та послідовність оліго (dT) для RT [30]. Процес зворотної транскрипції відбувається в капсульованих краплях, і їх синтезовані кДНК об'єднуються, ампліфікуються ПЛР і потім піддаються секвенуванню наступного покоління.
2.2. Аналіз scRNA-Seq визначає різні молекулярні підтипи стромальних фібробластів молочної залози у Brca1 -/-; p53 +/− миші
Одноклітинна послідовність зчитування РНК, необроблені дані, обробляли через конвеєр аналізу Cell Ranger для формування звітів про контроль якості scRNA-seq (рис. S1 – S4) та трьох наборів необроблених даних (штрих-код клітини, ознака гена та матриця підрахунку) для кожної зразки клітини (рис. 1 В) [31]. Як показано у звітах контролю якості, понад 600 клітин на зразок (RD-1: 1138, RD-2: 686, HFD-1: 1165, HFD-2: 903) були успішно секвенувані (Рисунки S1 – S4). Середні показники загальної кількості виявлених генів та медіани генів на клітину для цих чотирьох клітинних зразків складають відповідно 15 553 ± 309 та 1492 ± 257. Середній відсоток прочитаних, впевнено відображених на транскриптомі миші, становить 66,05% ± 7,69%. Ми провели аналіз scRNA-seq цих трьох наборів даних (клітинний штрих-код, ознака гена та матриця підрахунку) для кожної зразки клітини, використовуючи інструмент біопровідника на основі R, який називається Seurat (Рисунок 1 C) [32,33]. Оскільки для кожної групи дієт існувало два набори даних scRNA-seq, ми використали Seurat, щоб інтегрувати їх разом перед аналізом зменшення розмірності [33]. Завдяки аналізу з використанням алгоритмів зменшення розмірності, побудованих в Seurat, було проведено аналіз основних компонентів (PCA; рис. 2 А) та розподілене t-розподілене стохастичне сусідство (t-SNE; рис. 2 В) для класифікації типів клітин (рис. 2). Діаграми t-SNE для груп HFD та RD показують 14 різних кластерних клітин (Рисунок 2 B), що вказує на наявність принаймні 14 різних типів клітин у відсортованих зразках клітин.
Зменшення розмірності та аналіз теплової карти даних послідовності одноклітинної РНК (scRNA). (A) Аналіз ділянки PCA даних scRNA-seq. (B) Аналіз ділянки t-SNE даних scRNA-seq. Імена клітинних типів коментуються на графіках на основі ідентифікованих маркерних генів, характерних для клітинного типу. (C.) Теплова карта найкращих диференційовано експресованих генів у кожному підтипі стромальних фібробластів (Топ 12 генів для T1 – T4 і Топ 6 генів для T5).
Проаналізувавши окремий кластер клітин за допомогою алгоритму пошуку генних маркерів, побудованого в Seurat, ми визначили специфічні для клітин типу генні маркери для класифікації типів клітин. Експресія ідентифікованих генних маркерів у цих 14 клітинних кластерах відображалася на ділянках скрипки (Vlnplots; малюнок 3) та ділянках генних ознак t-SNE (рисунок 4). На основі цих ідентифікованих для клітин типу генетичних маркерів, показаних на малюнках 3 та 4, ми змогли присвоїти відомі імена клітинних типів 13 із 14 кластерних кластерів (малюнок 2 B), включаючи стромальні фібробласти (Vim +; 5 різних клітин кластери), Т-клітини пам'яті (Cd3d +, Lef1 +, Crem +), наївні Т-клітини (Cd3d +, Lef1 +, Продати +), В-клітини (Cd79a +), NK-клітини (Gzma +, Nkg7 +), монцити (Cd14 +, Tnip3 +, Retnla +, Cd68 +), макрофаги (Cd14 +, Retnla +, Cd68 +, Cd163 +, C1qb +), шкірні дендритні клітини (шкірні DC; Cd14 +, Clec4e +) [34] та дендритні клітини (DC; Siglech +) [35]. За допомогою аналізу scRNA-seq ми вперше виявили п’ять різних типів стромальних фібробластів, присутніх у Brca1 -/-; p53 +/− молочні залози, включаючи строму T1 (високий Tnfaip6), строму T2 (Igfbp5 +), строму T3 (високу червня), строму T4 (Rgcc +) і строму T5 (високу Klf2) (рисунок 2, малюнок 3 і рисунок 4).
Експресійний аналіз специфічних для клітин типу маркерних генів на основі даних scRNA-seq. Експресія цих маркерних генів була представлена в Vlnplots. Алгоритм аналізу диференціальної експресії генів, вбудований в пакет Seurat, був використаний для ідентифікації маркерних генів, переважно або однозначно експресованих у кожному типі клітин.
- Прибито! Зірка Жака Торреса виявляє 60 фунтів
- Наомі Кемпбелл розповідає, що їсть лише одну їжу на день PerthNow
- Наталі Дормер виявляє вражаючий абс на жінок; s Охорона здоров'я
- Національний місяць харчування, як дотримуватись дієти, збагаченої суперпродуктами, розкрито експертами - NDTV Food
- Расові відмінності у харчуванні міських неблагополучних чорношкірих та білих людей виявляють стійкість до