Характеристика генетичної втрати функції Fus у даніо
Світлана Лебедєва
Інститут молекулярної біології, Майнц, Німеччина
Антоніо М. де Ісус Домінгуес
Інститут молекулярної біології, Майнц, Німеччина
Фальк Баттер
Інститут молекулярної біології, Майнц, Німеччина
Рене Ф. Кеттінг
Інститут молекулярної біології, Майнц, Німеччина
Пов’язані дані
АНОТАЦІЯ
РНК-зв’язуючий білок FUS бере участь у транскрипції, альтернативному сплайсингу нейрональних генів та репарації ДНК. Мутації в FUS пов'язані з нейродегенеративними захворюваннями людини, такими як ALS (бічний аміотрофічний склероз). Ми генетично порушили фуз у даніо (Danio rerio) за допомогою системи CRISPR-Cas9. Тварини, що нокаутують фуз, плодючі та не виявляли жодного характерного фенотипу. Мутація грибка викликає незначні зміни в експресії генів на рівні транскриптома та протеома в мозку дорослого. Ми спостерігали значний вплив генетичного фону на експресію генів та використання 3'UTR, що може замаскувати наслідки втрати Fus. На відміну від опублікованих fus morphants, материнські зиготичні fus-мутанти не виявляють мотонейрональної дегенерації та демонструють нормальну рухову активність.
Скорочення
Вступ
Результати
Алелі нокауту Fus у даніо
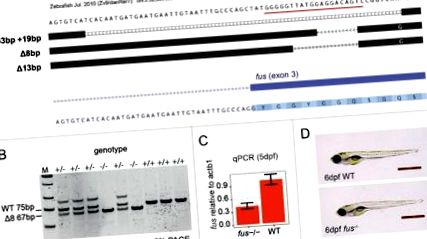
Ми використовували технологію CRISPR-Cas9 24, щоб зруйнувати ген fus данио-риби за допомогою однієї направляючої РНК, націленої на екзон 3 (рис. 1А). Всі наступні аналізи проводили на алелі, який видаляє 8 пар основ, в результаті чого відбувається зсув кадру та передчасний стоп-кодон (алель Δ8) (рис. 1А, Б). ІР-мРНК 88 все ще була у тварин, що нокаутували, хоча вона була приблизно в 2 рази менше в порівнянні з масовою мРНК (рис. 1С). Опубліковані антитіла, які використовувались для виявлення білка Fus даніо на Western blot 21, не змогли виявити Fus в ембріонах дикого типу в наших руках (не показано). Тому ми використовували мас-спектрометрію (МС) для того, щоб визначити, чи мРНК us8 злиття все ще здатна продукувати білок. Щоб мінімізувати можливий внесок материнського депонованого білка, ми проаналізували 5-денні ембріони з гетерозиготних поперек. Жодного пептиду, унікального для білка Fus, не було виявлено в жодній з 4 копій ембріонів fus -/-, тоді як 2 унікальних пептиду Fus були присутні у ¾ репліках братів і сестер дикого типу (табл. 1). Цей аналіз привів нас до висновку, що білок Fus не вироблявся, а алель fus Δ8 є мутацією втрати функції.
Генерування та перевірка дановидних риб, що нокаутують fus. (A) алелі фуза, генеровані CRISPR-Cas9. Знімок екрана з браузера геному UCSC показує вирівняні послідовності з гетерозиготних тварин F1. Цільовий сайт CRISPR у fus exon 3 підкреслено червоним кольором. (B) Приклад гелю PAGE для генотипування алелю Δ8. М = маркер низької молекулярної маси. Продукти дикого типу (75bp) та fus -/- (67bp) можуть бути дискриміновані. (C) Кількісне визначення мРНК fus щодо мРНК WT в ембріонах 5dpf. (D) Нормальна морфологія WT та fus -/- ембріона через 6 днів після запліднення (dpf). Шкала ваги 1мм.
Таблиця 1.
Ідентифікація Fus методом мас-спектрометрії (MS). Пептиди, унікальні для білка Fus, були ідентифіковані лише в аналізі РС ембріонів WT 5dpf та мозку дорослих, але не в нокауті Fus. Хрестик (х) означає, що пептид був ідентифікований у відповідному зразку. Внизу: Повна послідовність білка Fus, виявлені унікальні для Fus пептиди виділені жирним шрифтом та підкреслено.
| дикий тип | fus -/- | ||||||||
| Зразок | Послідовність | репл. 1 | репл. 2 | репл. 3 | репл. 4 | репл. 1 | репл. 2 | репл. 3 | репл. 4 |
| Ембріон | AAIDWFDGKDFNGNPIK | X | |||||||
| TGLPMINLYTDR | X | X | |||||||
| Мозок | AAIDWFDGKDFNGNPIK | X | X | X | X | ||||
| CSNPSCGNLNFSWR | X | X | X | ||||||
| TGLPMINLYTDR | X | ||||||||
MASNDYGQTSSHGYGGYGGQSGQSYSQPSAQNYSQQSYGGYNQSSESSSAPYNQGGYSSNYGQSQSGGYGSQAPSQGYSQSSQSYSSGGYSNTSQPPPAQSGGYSQQSSYSGYNQSSPASAPGGYSSSSQSSGYGQQQQQSGGGYGGSGGQSGGYGSSGGQSSGFGGSGGQHQSSQSGGGSYSPSPNYSSPPPQSYGQQSQYGQGGYNQDSPPMSGGGGGGGYGGQDGGYSQDGRGGRGRGGGFGGRGAGGFDRGGRGGPRGRGGMGMGDRGGFNKFGGPRDHGAGGPNMQEQDNSDNNTIFVQGLGDDYTVDSVADYFKQIGIIKVNKKTGLPMINLYTDRETGKLKGEATVSFDDPPSAKAAIDWFDGKDFNGNPIKVSFATRRAEFGRGGSSGGMRGGRGRGGPMGRGGFGGGRGGGGGGGGFQGNNGGGSGNGGGQQRAGDWKCSNPSCGNLNFSWRNECNQCKEPKPEGSGGGMSPMGGGFGGERGRSGFDRGGFRGRGGDRGGFRGGRGGDRGGFGPGKMDSRGDHRHDRRDRPY
Аналіз транскриптома та протеома мозку, що нокаутує фуз
Транскриптом і протеом злиття -/- мозок. (A) Дорослий. мозок данини від братів і сестер fus -/- або WT був використаний для одночасної кількісної оцінки мРНК шляхом секвенування РНК та білків за допомогою мас-спектрометрії. Всього було виміряно транскриптоми 5 фуз -/- та 4 ваг., Та протеоми 4 фуз -/- та 4 ват. Тварин. (B) Діаграма вулкана змін складок мРНК відображає зміну складки (на осі x) і значення р (на осі y). fus - це найбільш суттєво змінений ген на рівні мРНК (позначений червоним кольором) (C) Кількісне визначення протеому методом мас-спектрометрії без міток. Кількісне визначення без міток проводили, як описано раніше. 43 Середня експресія білків у мозку WT (вісь y) побудована проти експресії в fus -/- мозку (вісь x) за шкалою журналу. Білкам, не виявленим в одній із зразків, було присвоєно довільне значення 22. Fus є одним з найбільш виражених білків, які були виявлені у ВТ та відсутні у нокауті (позначено червоним).
Генетичний фон впливає на альтернативне використання 3'UTR
Під час аналізу даних РНК-секвенування ми помітили, що деякі гени демонструють значні зміни в експресії генів серед братів і сестер (біологічні копії), незалежно від генотипу fus (рис. S3). Риб даніо не можна підтримувати як інбредні лінії через інбридингову депресію26, а фон нашої лінії змішаний (AB × TU). Ми розсудили, що генетичні фонові ефекти можуть переважати над тонкими ефектами нокауту. Щоб вирішити цю проблему більш систематично, ми вирішили зосередитись на використанні альтернативних ділянок поліаденілювання, що є відносно простим для кількісного визначення та візуалізації. Ми використовували isoSCM 27 для нового виявлення ізоформ 3′UTR та кількісно визначали їх диференціальну експресію. Далі ми назвали однонуклеотидні поліморфізми (SNP) з даних RNA-Seq, використовуючи перевагу достатнього охоплення зчитування над 3'UTR. Дійсно, найвищі диференціальні ізоформи 3′UTR часто дотримувались розподілу SNP, а не генотипу fus (рис. 3). Таким чином, ми можемо виявити диференціальну експресію ізоформи в наших даних РНК-Seq, але, схоже, на використання ізоформи сильніше впливає місцевий генетичний фон, ніж експресія Fus.
Приклади альтернативного використання 3'UTR у різних генетичних фонах. Показані скріншоти з браузера геному IGV. Відповідними генами є slc37a4b, pomp та imp3. Для зручності доріжки покриття RNA-Seq показані синім лише для мінусової нитки. Викликані SNP, які були відфільтровані (див. Методи), представлені кольоровими смужками над генною доріжкою. Темно-сині - гомозиготні варіанти, темно-червоні смуги - гетерозиготні; гомозиготні посилання або відсутні дзвінки не відображаються. Трек isoSCM показує передбачувані альтернативні 3'кінці, викликані isoSCM. Шкала покриття RNA-Seq нормується до 1 000 000/(загальна кількість зчитувань) і однакова для всіх доріжок (зображена сірим кольором у квадратних дужках праворуч).
Ембріони з нокаутом Fus не мають дефектів мотонейронів
Морфологія і дотик мотонейрону викликали реакцію на втечу личинок, що вибивають фуз. (А) Конфокальні зображення стовбурів личинок 36 к.с., що експресують нейрон-специфічний DsRed. 28 Личинки нокауту Fus-матерів-зиготиків виявляють нормальну загальну морфологію первинного мотонейрону аксону, що не відрізняється від WT. Показана проекція максимальної інтенсивності конфокального стека; шкала - 100 мкм. (B) Дотик викликав реакцію втечі личинок 2dpf (48-52hpf). Побудовано відстань плавання в мм для окремих доріжок личинок. Значення Р отримано з тесту Крускала-Уолліса. n вказує кількість випробуваних личинок для кожної групи.
Обговорення
Ми використовували безпородних даніо (AB × TU) для генерування тварин, що нокаутують фус. Ця стратегія була вигідною, оскільки ембріони у фоновому режимі, ін'єктовані висококонцентрованим Cas9 та провідниковою РНК, показали погану виживаність. Однак ми спостерігали, що генетичний фон може сильно впливати на експресію генів та альтернативне використання ізоформи. Ці ефекти могли приховати незначні зміни вираження, спричинені втратою Fus. Крім того, наші дані свідчать про те, що ефекти ін’єкцій морфоліно можуть проявлятися по-різному залежно від генетичного фону ембріонів. Відповідно до цього, кілька моделей нокаутованих мишей FUS демонструють широкий спектр фенотипів, які варіюються від постнатальної летальності в більш інбредних лініях до дорослих, що вижили з легкими морфологічними та поведінковими дефектами в безпородній лінії. 18-20 Оскільки висока щільність SNP і безпородний характер даніо більше схожий на характер у людей, ніж у інбредних ліній мишей, даніо може представляти дуже корисну модель для вивчення ефекту Fus або будь-якого іншого білка, в контексті природних генетичних варіацій.
Сирі дані RNAseq зберігаються в GEO (> GSE85554). Дані протеоміки були передані до Консорціуму ProteomeXchange через сховище партнерів PRIDE з ідентифікатором набору даних PXD004876e.
Методи
Розширені методи можна знайти в Додатковому матеріалі.
Рибництво та штами
Рибу утримували, вирощували та влаштовували, як описано раніше. 35 Якщо не зазначено інше, у всіх експериментах використовували суміш штаму AB і TU. Усі експерименти проводились відповідно до німецького закону про добробут тварин (ліцензії 23 177–07/G 13–5–087 (нокаути CRISPR-Cas9), 23177–07/A 15–5–001 OES (затискачі хвостових плавників)). Нокаути CRISPR-Cas9 були створені, як описано. 24
секвенування мРНК
Бібліотеки поліаденильованих РНК, специфічні для ланцюга, готували за допомогою набору для підготовки бібліотечних мереж TruSeq і послідовно розподіляли на Illumina HiSeq2000 із використанням протоколу зчитування 2 × 100 bp. Зчитування послідовності РНК накладали на геном даніо (Zv10) за допомогою вирівнювача STAR (версія 2.4.1d 36). Таблиця підрахунку прочитаних була створена за допомогою featureCounts (v. 1.4.6 37) з використанням анотації генів бази даних Ensembl (випуск 80). Диференціально експресовані гени називали за допомогою пакета R DESeq2. 38
Мас-спектрометрія
Підготовку та вимірювання зразків мас-спектрометрії проводили, як описано 39, за винятком 50 см колонки С18 та використовували 240-хвилинний градієнт РХ. Зразки аналізували за допомогою MaxQuant (версія 1.5.2.8 40) щодо наданої бази даних даніо Uniprot (39 559 записів) зі стандартними налаштуваннями, за винятком того, що значення LFQ базувалися на унікальних пептидах. Для аналізу даних інформацію про ідентифіковані пептиди було вилучено із таблиці доказів. На рівні білка середні значення LFQ були розраховані для кожного стану (WT, fus -/-) з файлу білкових груп та побудовані як графік розсіювання в R, використовуючи пакет ggplot2. 41
Конфокальна візуалізація
Стовбури данини були зображені на конфокальному мікроскопі Leica SP5. Максимальна проекція зображень була створена за допомогою програмного забезпечення Фіджі. 42
Ін’єкції морфоліно
Fus morpholino (GGCCATAATCATTTGACGCCATGTT) 21 та 5-невідповідні контрольний морфоліно (GCCCATAATGATTTCACGGCATCTT) були від Genetools. Приблизно 1 нл краплі 1 мМ Fus morpholino вводили в ембріони 1-клітинної стадії.
- Повна стаття 167 Фізико-хімічна характеристика амілаз L
- Генетична взаємодія з контрацептивами може спричинити збільшення ваги
- Снідайте великим сніданком, щоб схуднути Ця стара харчова приказка не; т підкріплено наукою Генетична
- Розробка та характеристика нового гідрогелю на основі галактоманнану та κ-карагенану -
- Калькулятор FFMI Розрахуйте свій генетичний м’язовий потенціал